Quem não gostou de colecionar construtores na infância? Ainda me lembro dessa caixa vermelha com um monte de peças de metal, ferramentas e um mar de resultados possíveis, se houvesse fantasia, tempo e desejo. LEGO também não deve ser esquecido, embora aqui tudo fosse um pouco mais simples e muito mais colorido. Mas o mais complicado de ambos são os projetistas de nanoestruturas baseadas no origami de DNA. Até agora, todos os "detalhes" dessas estruturas eram modelados manualmente, o que demandava muito tempo e esforço. Imagine que você precisa criar todas as peças LEGO antes de montá-las em um robô gigante com lasers, motores a jato e uma metralhadora no seu ombro. Mas algo que as lembranças da infância nos levaram à estepe errada.
Brincadeiras à parte. Hoje conheceremos o algoritmo que permite criar automaticamente origens de DNA de uma forma bastante diversa. Anteriormente, a alteração do formato das cadeias de DNA para o necessário era realizada manualmente, o que limitava bastante as possibilidades de tal procedimento. O mesmo algoritmo permite criar partes de DNA de designer automaticamente, o que permite usá-las para a formação de nanoestruturas bidimensionais e tridimensionais. Além disso, esse algoritmo está disponível para todos. Um relatório da equipe de pesquisa nos ajudará a descobrir o que funciona e como. Vamos lá
Base de estudoO uso do DNA como material de construção em nanoestruturas de uma ampla gama de aplicações nos últimos anos tornou-se o objetivo de muitos estudos e experiências. Muitos deles alcançaram excelentes resultados, mas, como sabemos, não há limite para a perfeição. Como exemplo, os pesquisadores citam estruturas de DNA monodispersas em uma escala de megadalt com um alto coeficiente de coagulação, que foi realizado usando longas estruturas de ssDNA (DNA de fita simples) - DNA de fita simples / de fita simples. No entanto, nesse caso, é o comprimento das cadeias de DNA que é a principal limitação no tamanho de um único DNA de origami. Essa limitação pode ser contornada pela formação de uma estrutura que consiste em vários origami de uma só vez.
Também já sabemos (
Tic-tac-toe: uma demonstração do processo controlado de reconfiguração de estruturas de DNA ) que algumas estruturas de DNA podem ser criadas usando blocos de DNA, o que permite formar uma geometria complexa dessa mesma estrutura.
Mas, novamente, tudo se resume ao fato de o processo ser realizado manualmente, e isso é longo e sombrio. Do que os cientistas gostam? É isso mesmo, automação ou pelo menos automação parcial. Ferramentas semelhantes já estão disponíveis (um exemplo de cientistas - caDNAno ou Tiamat), no entanto, esses programas têm suas desvantagens. Em primeiro lugar, a criação manual do quadro. Em segundo lugar, as limitações dos algoritmos acima na questão da sistematização do design da geometria da estrutura.
Em suma, neste artigo, os pesquisadores estabeleceram o objetivo de criar um algoritmo que fará quase tudo sozinho. Assim, qualquer pessoa pode definir apenas os parâmetros do invólucro externo da estrutura desejada, e o programa preencherá a parte interna independentemente com todos os fios, grampos, seqüências de DNA necessários e muito mais. Você também pode executar o processo na ordem inversa - defina todas as "partes internas", por assim dizer, a grade da estrutura e o algoritmo criará a estrutura correta e mais ideal ao seu redor.
Este programa de código aberto (PERDIX) está disponível para absolutamente todos, pelos quais agradecemos aos pesquisadores. Assim, qualquer um pode não apenas "brincar" com o próprio programa, mas também, possivelmente, fazer algumas alterações aprimoradas em seu código. Onde baixá-lo e como iniciá-lo, consideraremos no final do artigo.
E agora vamos ver exemplos da aplicação deste programa que a equipe de pesquisa nos forneceu.
Resultados da validação do PERDIX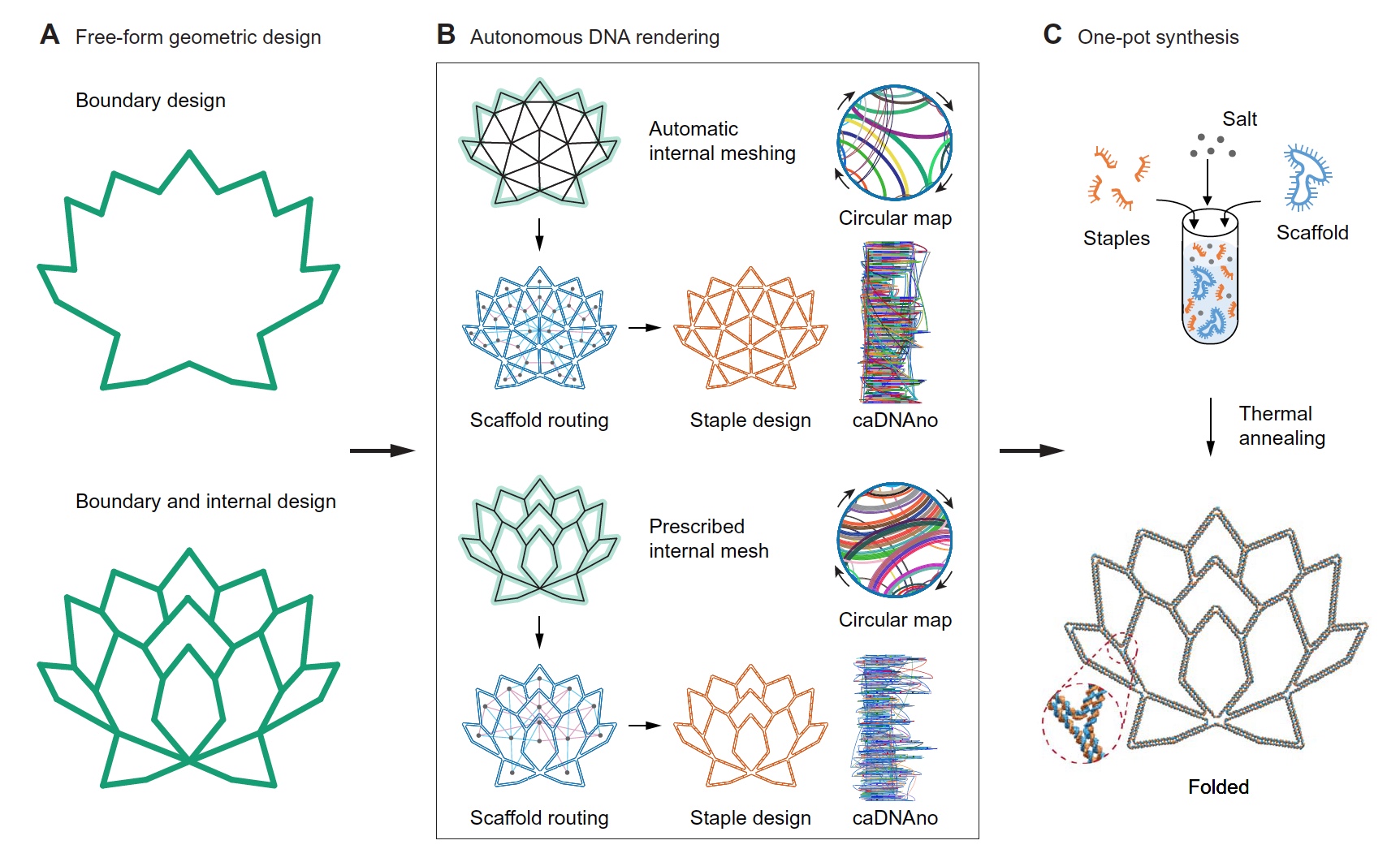 Imagem Nº 1
Imagem Nº 1Como dados de entrada, é usada a imagem da borda da estrutura desejada (
1A , acima) ou uma imagem com detalhes geométricos mais precisos (
1A , abaixo). Além disso, o próprio programa preenche o espaço interno.
Quando apenas o “contorno” é especificado, a geometria interna da grade da estrutura é formada por triângulos usando o programa de código aberto DistMesh, que requer a introdução apenas da densidade da grade como parâmetro necessário.
Quando a forma e as linhas geométricas são especificadas (
1A , inferior), Shapely é usado - um pacote Python para manipular e analisar objetos geométricos planos. Shapely gera uma malha poligonal, na qual suas linhas e seus pontos de interseção formam a geometria necessária do quadro.
Além disso, após determinar a forma desejada, era necessário definir o comprimento nominal da nervura, que deveria ser ≥ 38 pb (bases emparelhadas) ou 12,58 nm. Assim, pelo menos dois cruzamentos de emparelhamento por aresta estarão definitivamente presentes na estrutura necessária.
Em seguida, todas as linhas de destino da estrutura futura são transformadas em arestas DX, nas quais todos os vértices são pontos de conexão de grade múltipla (
1B ). Seguindo os princípios da teoria dos grafos, a estrutura esquelética ideal é formada automaticamente, após o que o processo de atribuição de uma sequência de grampos de DNA adicionais é concluído. O processo de edição manual dos dados de saída (resultados) é realizado por meio do arquivo caDNAno gerado.
Uma característica muito importante de seu sistema, os cientistas chamam o fato de que as costelas não precisam ser iguais a um número inteiro de hélices de DNA B duplo (10,5 pb). E isso permite que você obtenha mais liberdade no design da geometria da estrutura.
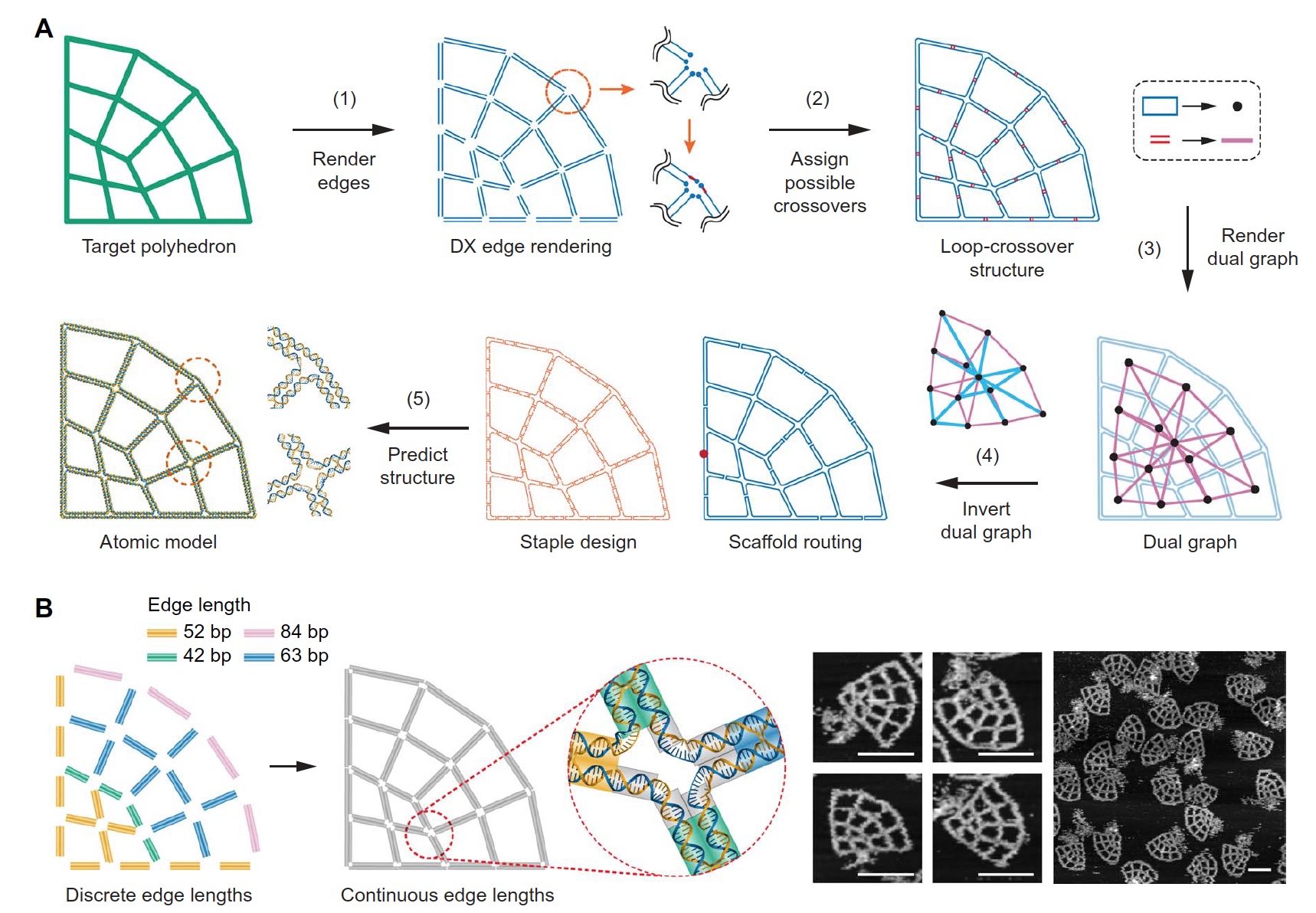 Imagem No. 2
Imagem No. 2Para executar com rapidez e precisão a parte automática do processo de formação da geometria da estrutura, todas as arestas devem primeiro ser convertidas em motivos DX renderizando cada aresta da geometria de destino usando duas linhas de armação antiparalelas (
2A ). Em seguida, eles são combinados em um único vértice, o que torna cada aresta parte de um loop. E isso, por sua vez, leva à formação de um único loop externo grande, consistindo em pequenos loops. O próximo passo é encontrar todas as opções de cruzamento possíveis entre os loops adjacentes, ou seja, a formação de uma estrutura de interseção de loop.
A terceira etapa deste processo é representar todas as estruturas resultantes como um conjunto de nós e todos os cruzamentos como arestas da estrutura futura.
Em seguida, é calculada a árvore de abrangência do gráfico duplo, após o que é convertida em uma estrutura ssDNA. Este processo é totalmente automático. E, finalmente, um modelo estrutural tridimensional é gerado no nível atômico.
A Figura
2B mostra duas opções para a formação da geometria necessária da estrutura: com comprimento discreto da aresta ou com comprimento contínuo da aresta. A segunda opção permite criar uma estrutura com um comprimento de borda arbitrário, o que, por sua vez, possibilita a obtenção da maior coagulação. Essas variações da estrutura da geometria foram verificadas usando um microscópio de força atômica de varredura (imagens à direita na imagem
2B ).
 Imagem No. 3
Imagem No. 3Os pesquisadores também estabeleceram o objetivo de examinar mais detalhadamente aspectos da geometria alvo, como rigidez mecânica, precisão da forma, tamanho da estrutura, tamanho da malha interna e tipos de seus elementos. Também foi necessário entender o quão importante os N-ramos desempenham, ou seja, o número de conexões entre as arestas e, como resultado, a complexidade geral da estrutura. A análise dos dados AFM mostrou que estruturas bidimensionais com um grande número de ramificações e um comprimento de borda mais longo (devido a nucleotídeos não emparelhados) são obtidas bastante bem formadas, e a heterogeneidade de partículas em sua estrutura é mínima.
A grade interna da estrutura tinha três opções: triângulos com a mesma direção, quadrados e triângulos com direções diferentes (
3C ). Foi o modelo com triângulos que mostrou a maior precisão na formação da geometria necessária, em contraste com o modelo com quadrados. A presença de direções mistas no modelo triangular, por assim dizer, possibilitou obter ramos N mais simétricos e uma forma mais precisa (menos distorcida).
Os cientistas observam que a análise da malha interna da estrutura simulada é extremamente importante para entender a rigidez mecânica dessa estrutura. E é precisamente o modelo descrito acima que é ideal para criar estruturas suficientemente fortes, precisas (em termos de cumprimento de parâmetros especificados) e estruturas flexíveis.
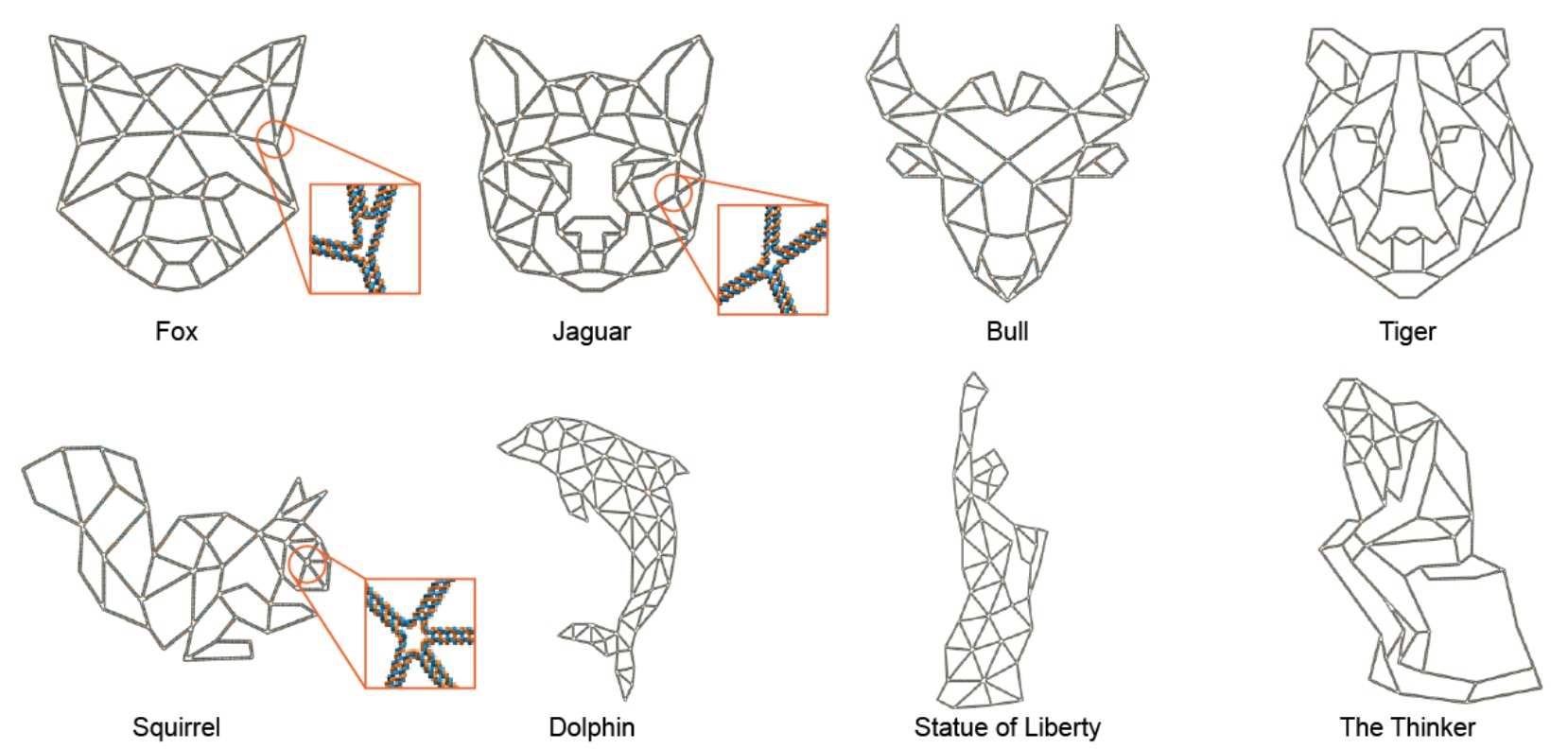
 Imagem No. 4
Imagem No. 4Obviamente, os cientistas não deixam de ter um senso de beleza, por assim dizer. Portanto, eles decidiram demonstrar as possibilidades de seu desenvolvimento, projetando estruturas de 15 formas diferentes, mais precisamente com diferentes grades e armações internas. Podemos ver as opções na imagem acima: aqui estão os quadrados "usuais", um quarto de círculo e até um lótus.
Software de código abertoComo mencionei anteriormente, o trabalho dos pesquisadores é aberto a todos no link (), onde você pode encontrar os elementos necessários do programa (MATLAB, Python 2.7 e Shapely 1.6.4).
Quanto ao algoritmo para a formação da geometria das estruturas de DNA-origami, as instruções para download e execução são apresentadas abaixo em formato de vídeo. Vou esconder todos os vídeos embaixo do spoiler para não esticar o artigo.
Vídeo # 1: lançamento do PERDIXO link para baixar os arquivos necessários que estavam no vídeo não funciona agora. Aqui está uma alternativa -
link para download .
Vídeo nº 2: projetando o quadro (perímetro) de uma estrutura no PERDIX
Vídeo # 3: projetando a estrutura e a estrutura interna no PERDIX
Vídeo nº 4: modelos atômicos (grades diferentes)
Vídeo nº 5: modelos atômicos (ramos N)
Vídeo nº 6: modelos atômicos (modelo L)
Vídeo nº 7: modelos atômicos ("ramo" curvo)
Para um conhecimento mais detalhado deste estudo e de seus resultados, recomendo fortemente que você analise o
relatório do grupo de pesquisa e
os materiais adicionais a ele.
EpílogoEste estudo teve como objetivo simplificar um processo bastante demorado e demorado. E deu certo. O algoritmo PERDIX permite criar estruturas com geometrias muito diferentes, enquanto define os parâmetros mínimos necessários. O DNA tem sido um objeto de estudo para os cientistas, não apenas como parte de toda a vida e veículo de informação, mas também como uma opção possível para a fundação de futuras tecnologias. Tais trabalhos permitem compreender com mais detalhes (e claramente) quão amplas são as possibilidades de nanoestruturas criadas com base no origami de DNA.
O segundo momento agradável deste estudo, pelo menos para mim, é que o algoritmo está disponível para todos. Qualquer um pode usá-lo, qualquer um pode melhorá-lo. Ao dar acesso total ao relatório e ao próprio software, os cientistas não apenas contribuem para a popularização de seu campo científico, mas também para a ciência como tal.
Conheci artigos nos quais os cientistas eram bastante críticos sobre o sistema pago de acesso a relatórios, argumentando que isso coloca uma barreira adicional entre o conhecimento e as pessoas comuns. A situação nesta questão é muito ambígua e controversa, porque todos deveriam ganhar dinheiro (publicações científicas, grupos de pesquisa e sites científicos), mas o alto custo do acesso a alguns estudos os torna inacessíveis aos amantes comuns da ciência, estudantes e até alguns professores e figuras científicas. Como eu disse, a questão é ambígua, portanto, não a tocaremos muito. De qualquer forma, fico feliz que nossos heróis de hoje tenham nos dado acesso total ao seu trabalho, que merece não apenas atenção, mas também admiração franca.
Obrigado pela atenção, continuem curiosos e tenham uma boa semana de trabalho, pessoal.
Obrigado por ficar conosco. Você gosta dos nossos artigos? Deseja ver materiais mais interessantes? Ajude-nos fazendo um pedido ou recomendando a seus amigos, um
desconto de 30% para os usuários da Habr em um análogo exclusivo de servidores básicos que inventamos para você: Toda a verdade sobre o VPS (KVM) E5-2650 v4 (6 núcleos) 10GB DDR4 240GB SSD 1Gbps da US $ 20 ou como dividir o servidor? (as opções estão disponíveis com RAID1 e RAID10, até 24 núcleos e até 40GB DDR4).
VPS (KVM) E5-2650 v4 (6 núcleos) 10 GB DDR4 240 GB SSD de 1 Gbps até a primavera, gratuitamente, ao pagar por meio ano, você pode fazer o pedido
aqui .
Dell R730xd 2 vezes mais barato? Somente nós temos
2 TVs Intel Dodeca-Core Xeon E5-2650v4 128GB DDR4 6x480GB SSD 1Gbps 100 a partir de US $ 249 na Holanda e nos EUA! Leia sobre
Como criar um prédio de infraestrutura. classe usando servidores Dell R730xd E5-2650 v4 custando 9.000 euros por um centavo?