Olá pessoal!
No
último artigo, descobrimos como as árvores de decisão são organizadas e, do zero, implementamos
algoritmo de construção, otimizando e melhorando simultaneamente. Neste artigo, implementaremos o algoritmo de aumento de gradiente e, no final, criaremos nosso próprio XGBoost. A narração seguirá o mesmo padrão: escrevemos um algoritmo, descrevemos e resumimos comparando os resultados do trabalho com análogos do Sklearn.
Neste artigo, a ênfase também será colocada na implementação em código; portanto, é melhor ler toda a teoria juntos em outra (por exemplo,
no curso ODS ) e, já com o conhecimento da teoria, você pode prosseguir para este artigo, pois o tópico é bastante complicado.
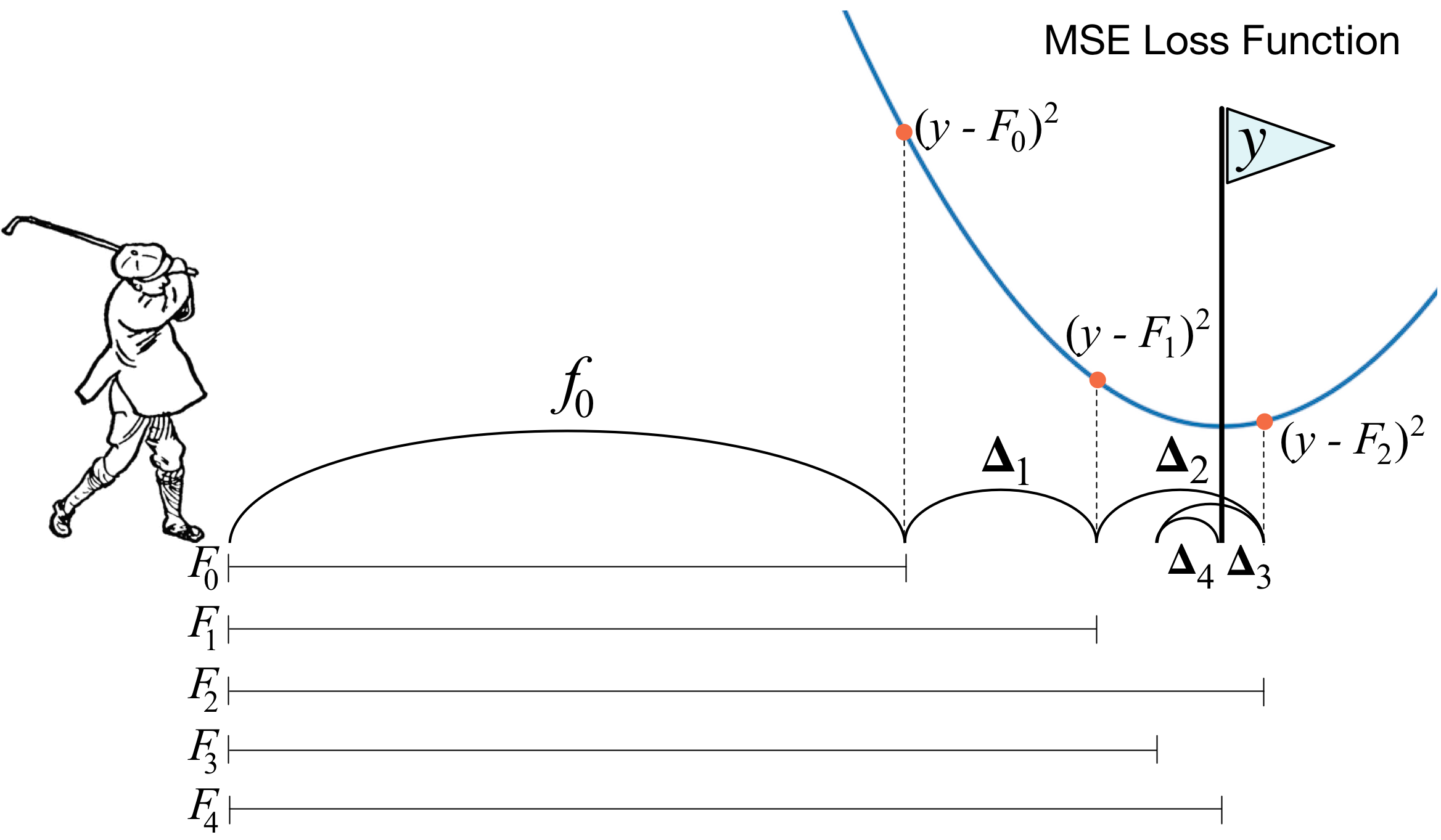
O que é aumento de gradiente? A imagem de um jogador de golfe descreve perfeitamente a idéia principal. Para empurrar a bola para dentro do buraco, o jogador de golfe faz cada próximo golpe levando em consideração a experiência de tacadas anteriores - para ele, essa é uma condição necessária para colocar a bola no buraco. Se for muito rude (não sou mestre em golfe :)), a cada novo golpe, a primeira coisa que um jogador observa é a distância entre a bola e o buraco após o golpe anterior. E a principal tarefa é reduzir essa distância com o próximo golpe.
O impulso é construído de maneira muito semelhante. Primeiro, precisamos introduzir a definição de "buraco", ou seja, a meta pela qual lutaremos. Em segundo lugar, precisamos aprender a entender de que lado precisamos vencer com um clube para nos aproximarmos do objetivo. Em terceiro lugar, levando em consideração todas essas regras, é necessário criar a seqüência correta de tacadas para que cada uma delas subseqüente reduza a distância entre a bola e o buraco.
Agora, damos uma definição um pouco mais rigorosa. Introduzimos o modelo de votação ponderada:
h(x)= sumni=1biai,x emX,bi emR
Aqui
X É o espaço do qual tiramos objetos,
bi,ai - este é o coeficiente na frente do modelo e o próprio modelo, ou seja, a árvore de decisão. Suponha que já em alguma etapa, usando as regras descritas, fosse possível adicionar à composição
T−1 algoritmo fraco. Para aprender a entender que tipo de algoritmo deve estar na etapa
T , apresentamos a função de erro:
err(h)= sumNj=1L( sumT−1i=1aibi(xj)+bTaT(xj)) rightarrowminaT,bTAcontece que o melhor algoritmo será aquele que pode minimizar o erro recebido nas iterações anteriores. E como o aumento é gradiente, essa função de erro deve necessariamente ter um vetor antigradiente ao longo do qual você pode se mover em busca de um mínimo. Isso é tudo!
Imediatamente antes da implementação, adicionarei algumas palavras sobre como tudo será organizado conosco. Como no artigo anterior, tomamos o MSE como uma perda. Vamos calcular seu gradiente:
mse(y,prever)=(y−prever)2 nablaprevermse(y,prever)=prever−y
Assim, o vetor antigradiente será igual a
y−prever . Na etapa
i consideramos os erros do algoritmo obtido em iterações anteriores. Em seguida, treinamos nosso novo algoritmo nesses erros e o adicionamos ao nosso conjunto com um sinal de menos e algum coeficiente.
Agora vamos começar.
1. Implementação da classe usual de aumento de gradiente
import pandas as pd import matplotlib.pyplot as plt import numpy as np from tqdm import tqdm_notebook from sklearn import datasets from sklearn.metrics import mean_squared_error as mse from sklearn.tree import DecisionTreeRegressor import itertools %matplotlib inline %load_ext Cython %%cython -a import itertools import numpy as np cimport numpy as np from itertools import * cdef class RegressionTreeFastMse: cdef public int max_depth cdef public int feature_idx cdef public int min_size cdef public int averages cdef public np.float64_t feature_threshold cdef public np.float64_t value cpdef RegressionTreeFastMse left cpdef RegressionTreeFastMse right def __init__(self, max_depth=3, min_size=4, averages=1): self.max_depth = max_depth self.min_size = min_size self.value = 0 self.feature_idx = -1 self.feature_threshold = 0 self.left = None self.right = None def fit(self, np.ndarray[np.float64_t, ndim=2] X, np.ndarray[np.float64_t, ndim=1] y): cpdef np.float64_t mean1 = 0.0 cpdef np.float64_t mean2 = 0.0 cpdef long N = X.shape[0] cpdef long N1 = X.shape[0] cpdef long N2 = 0 cpdef np.float64_t delta1 = 0.0 cpdef np.float64_t delta2 = 0.0 cpdef np.float64_t sm1 = 0.0 cpdef np.float64_t sm2 = 0.0 cpdef list index_tuples cpdef list stuff cpdef long idx = 0 cpdef np.float64_t prev_error1 = 0.0 cpdef np.float64_t prev_error2 = 0.0 cpdef long thres = 0 cpdef np.float64_t error = 0.0 cpdef np.ndarray[long, ndim=1] idxs cpdef np.float64_t x = 0.0
class GradientBoosting(): def __init__(self, n_estimators=100, learning_rate=0.1, max_depth=3, random_state=17, n_samples = 15, min_size = 5, base_tree='Bagging'): self.n_estimators = n_estimators self.max_depth = max_depth self.learning_rate = learning_rate self.initialization = lambda y: np.mean(y) * np.ones([y.shape[0]]) self.min_size = min_size self.loss_by_iter = [] self.trees_ = [] self.loss_by_iter_test = [] self.n_samples = n_samples self.base_tree = base_tree def fit(self, X, y): self.X = X self.y = y b = self.initialization(y) prediction = b.copy() for t in tqdm_notebook(range(self.n_estimators)): if t == 0: resid = y else:
Agora, construiremos a curva de perda no conjunto de treinamento para garantir que a cada iteração realmente tenhamos uma diminuição.
GDB = GradientBoosting(n_estimators=50) GDB.fit(X,y) x = GDB.predict(X) plt.grid() plt.title('Loss by iterations') plt.plot(GDB.loss_by_iter)

2. Ensacamento sobre árvores decisivas
Bem, antes de compararmos os resultados, vamos falar sobre o procedimento de
empacotar árvores.
Tudo é simples aqui: queremos nos proteger da reciclagem e, portanto, com a ajuda da amostragem com retorno, calcularemos a média de nossas previsões para não gerar emissões acidentalmente (por que isso funciona - leia melhor o link).
class Bagging(): ''' Bagging - . ''' def __init__(self, max_depth = 3, min_size=10, n_samples = 10):
Bem, agora como algoritmo básico, podemos usar não apenas uma árvore, mas ensacá-las - então, novamente, nos protegeremos da reciclagem.
3. Resultados
Compare os resultados dos nossos algoritmos.
from sklearn.model_selection import KFold import matplotlib.pyplot as plt from sklearn.ensemble import GradientBoostingRegressor as GDBSklearn import copy def get_metrics(X,y,n_folds=2, model=None): kf = KFold(n_splits=n_folds, shuffle=True) kf.get_n_splits(X) er_list = [] for train_index, test_index in tqdm_notebook(kf.split(X)): X_train, X_test = X[train_index], X[test_index] y_train, y_test = y[train_index], y[test_index] model.fit(X_train,y_train) predict = model.predict(X_test) er_list.append(mse(y_test, predict)) return er_list data = datasets.fetch_california_housing() X = np.array(data.data) y = np.array(data.target) er_boosting = get_metrics(X,y,30,GradientBoosting(max_depth=3, n_estimators=40, base_tree='Tree' )) er_boobagg = get_metrics(X,y,30,GradientBoosting(max_depth=3, n_estimators=40, base_tree='Bagging' )) er_sklearn_boosting = get_metrics(X,y,30,GDBSklearn(max_depth=3,n_estimators=40, learning_rate=0.1)) %matplotlib inline data = [er_sklearn_boosting, er_boosting, er_boobagg] fig7, ax7 = plt.subplots() ax7.set_title('') ax7.boxplot(data, labels=['Sklearn Boosting', 'Boosting', 'BooBag']) plt.grid() plt.show()
Recebido:
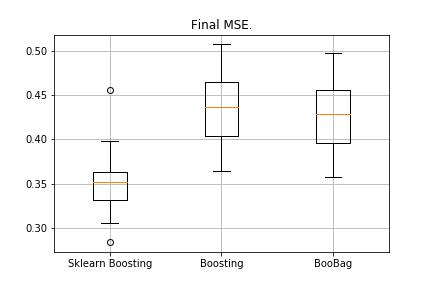
Ainda não podemos derrotar o analógico do Sklearn, porque novamente não levamos em consideração muitos parâmetros usados
neste método . No entanto, vemos que o ensacamento ajuda um pouco.
Não vamos nos desesperar e passar a escrever o XGBoost.
4. XGBoost
Antes de ler mais, recomendo fortemente que você se familiarize com o próximo
vídeo , o que explica muito bem a teoria.
Lembre-se de qual erro minimizamos no impulso normal:
err(h)= sumNj=1L( sumT−1i=1aibi(xj)+bTaT(xj))
O XGBoost adiciona explicitamente regularização a esta funcionalidade de erro:
err(h)= sumNj=1L( sumT−1i=1aibi(xj)+bTaT(xj))+ sumTi=1 ômega(ai)
Como considerar essa funcionalidade? Primeiro, nós o aproximamos com a ajuda de uma série de Taylor de segunda ordem, onde o novo algoritmo é considerado como um incremento ao longo do qual iremos mover e, em seguida, já pintamos dependendo do tipo de perda que temos:
f (x + \ delta x) \ thickapprox f (x) + f (x) '\ delta x + 0,5 * f (x)' '(\ delta x) ^É necessário determinar qual árvore consideraremos ruim e qual é boa.
Lembre-se de que princípio a
regressão é construída com
L2 -regularização - quanto mais normais os valores dos coeficientes antes da regressão, pior é, portanto, necessário que sejam tão pequenos quanto possível.
No XGBoost, a idéia é muito semelhante: a árvore é multada se a soma da norma dos valores nas folhas for muito grande. Portanto, a complexidade da árvore é apresentada da seguinte maneira:
omega(a)= gamaZ+0,5∗ sumZi=1w2i
w - valores nas folhas,
Z - número de folhas.
Existem fórmulas de transição no vídeo, não as exibiremos aqui. Tudo se resume ao fato de escolhermos uma nova partição, maximizando o ganho:
Gain= fracG2lS2l+ lambda+ fracG2rS2r+ lambda− frac(Gl+Gr)2S2l+S2r+ lambda− gamma
Aqui
gama, lambda Os parâmetros numéricos de regularização e
Gi,Si - as somas correspondentes da primeira e segunda derivadas para esta partição.
É isso, a teoria é bem resumida, os links são dados, agora vamos falar sobre quais serão os derivados se trabalharmos com o MSE. É simples:
mse(y,prever)=(y−prever)2 nablaprevermse(y,prever)=prever−y nabla2prevermse(y,prever)=1Quando vamos calcular a quantidade
Gi,Si , basta adicionar ao primeiro
prever−y e o segundo é apenas a quantidade.
%%cython -a import numpy as np cimport numpy as np cdef class RegressionTreeGain: cdef public int max_depth cdef public np.float64_t gain cdef public np.float64_t lmd cdef public np.float64_t gmm cdef public int feature_idx cdef public int min_size cdef public np.float64_t feature_threshold cdef public np.float64_t value cpdef public RegressionTreeGain left cpdef public RegressionTreeGain right def __init__(self, int max_depth=3, np.float64_t lmd=1.0, np.float64_t gmm=0.1, min_size=5): self.max_depth = max_depth self.gmm = gmm self.lmd = lmd self.left = None self.right = None self.feature_idx = -1 self.feature_threshold = 0 self.value = -1e9 self.min_size = min_size return def fit(self, np.ndarray[np.float64_t, ndim=2] X, np.ndarray[np.float64_t, ndim=1] y): cpdef long N = X.shape[0] cpdef long N1 = X.shape[0] cpdef long N2 = 0 cpdef long idx = 0 cpdef long thres = 0 cpdef np.float64_t gl, gr, gn cpdef np.ndarray[long, ndim=1] idxs cpdef np.float64_t x = 0.0 cpdef np.float64_t best_gain = -self.gmm if self.value == -1e9: self.value = y.mean() base_error = ((y - self.value) ** 2).sum() error = base_error flag = 0 if self.max_depth <= 1: return dim_shape = X.shape[1] left_value = 0 right_value = 0
Um pequeno esclarecimento: para tornar as fórmulas nas árvores com ganho mais bonitas, treinamos o alvo com um sinal de menos.
Modificamos levemente nosso impulso, adaptamos alguns parâmetros. Por exemplo, se notarmos que a perda começou a atingir um platô, diminuímos a taxa de aprendizado e aumentamos max_depth para os estimadores a seguir. Também adicionaremos uma nova ensacadeira - agora aumentaremos sobre as ensacadas das árvores com ganho:
class Bagging(): def __init__(self, max_depth = 3, min_size=5, n_samples = 10): self.max_depth = max_depth self.min_size = min_size self.n_samples = n_samples self.subsample_size = None self.list_of_Carts = [RegressionTreeGain(max_depth=self.max_depth, min_size=self.min_size) for _ in range(self.n_samples)] def get_bootstrap_samples(self, data_train, y_train): indices = np.random.randint(0, len(data_train), (self.n_samples, self.subsample_size)) samples_train = data_train[indices] samples_y = y_train[indices] return samples_train, samples_y def fit(self, data_train, y_train): self.subsample_size = int(data_train.shape[0]) samples_train, samples_y = self.get_bootstrap_samples(data_train, y_train) for i in range(self.n_samples): self.list_of_Carts[i].fit(samples_train[i], samples_y[i].reshape(-1)) return self def predict(self, test_data): num_samples = test_data.shape[0] pred = [] for i in range(self.n_samples): pred.append(self.list_of_Carts[i].predict(test_data)) pred = np.array(pred).T return np.array([np.mean(pred[i]) for i in range(num_samples)])
class GradientBoosting(): def __init__(self, n_estimators=100, learning_rate=0.2, max_depth=3, random_state=17, n_samples = 15, min_size = 5, base_tree='Bagging'): self.n_estimators = n_estimators self.max_depth = max_depth self.learning_rate = learning_rate self.initialization = lambda y: np.mean(y) * np.ones([y.shape[0]]) self.min_size = min_size self.loss_by_iter = [] self.trees_ = [] self.loss_by_iter_test = [] self.n_samples = n_samples self.base_tree = base_tree
5. Resultados
Por tradição, comparamos os resultados:
data = datasets.fetch_california_housing() X = np.array(data.data) y = np.array(data.target) import matplotlib.pyplot as plt from sklearn.ensemble import GradientBoostingRegressor as GDBSklearn er_boosting_bagging = get_metrics(X,y,30,GradientBoosting(max_depth=3, n_estimators=150,base_tree='Bagging')) er_boosting_xgb = get_metrics(X,y,30,GradientBoosting(max_depth=3, n_estimators=150,base_tree='XGBoost')) er_sklearn_boosting = get_metrics(X,y,30,GDBSklearn(max_depth=3,n_estimators=150,learning_rate=0.2)) %matplotlib inline data = [er_sklearn_boosting, er_boosting_xgb, er_boosting_bagging] fig7, ax7 = plt.subplots() ax7.set_title('') ax7.boxplot(data, labels=['GdbSklearn', 'Xgboost', 'XGBooBag']) plt.grid() plt.show()
A imagem será a seguinte:
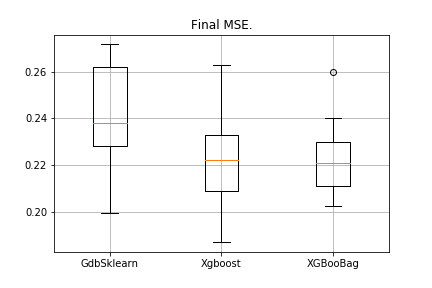
O XGBoost tem o erro mais baixo, mas o XGBooBag tem um erro mais cheio, o que é definitivamente melhor: o algoritmo é mais estável.
Isso é tudo. Eu realmente espero que o material apresentado em dois artigos seja útil e que você possa aprender algo novo por si mesmo. Agradeço especialmente a Dmitry pelo feedback abrangente e pelo código-fonte, a Anton pelo conselho e a Vladimir pelas tarefas difíceis de estudar.
Todo o sucesso!