1. Introdução
O artigo é destinado a quem já se interessou pela questão do que está acontecendo dentro da rede neural artificial
( RNA) . Agora quase todo mundo pode desenvolver sua própria RNA usando as bibliotecas prontas disponíveis na maioria das linguagens de programação. Neste artigo, tentarei mostrar como o objeto (
Pattern ) parece exatamente, passando pelas camadas da RNA, desenvolvido e compilado usando a
biblioteca de aprendizado profundo do
Tensorflow com o suplemento
Keras .
Software usado
Os seguintes componentes são necessários (as versões especificadas para o meu caso):
- tensorflow 1.10.0
- keras 2.2.4
- matplotlib 2.2.0
- modul-os
- numpy1.14.3
Também é possível desenhar a arquitetura da rede, mas para isso é necessário instalar ferramentas de visualização, no meu caso, o
keras foi usado e no método
PLOT_PATTERN_PROCCESS(...)
estabelecer
PLOT_MODEL = True def PLOT_PATTERN_PROCCESS(model, pattern, FOLDER_TO_SAVE, grid_size=(3, 3), limit_size_layer=(15, 15), PLOT_MODEL=True):
Ideia principal
É necessário escolher um padrão (a passagem que observaremos), depois de escolher, a rede é dividida em camadas
tensoras . Em um ciclo da segunda para a última camada, uma nova rede é criada, onde a saída é o número da camada no ciclo e pulando o padrão, o resultado é uma matriz n-dimensional na saída da rede.
Implementação
Conectando bibliotecas
from keras.models import * from keras.layers import * import matplotlib.pyplot as plt import os import numpy as np
Métodos utilizados:
def PLOT_PATTERN_PROCCESS (modelo, padrão, FOLDER_TO_SAVE, grid_size = (3, 3), limit_size_layer = (15, 15), PLOT_MODEL = True): def PLOT_PATTERN_PROCCESS(model, pattern, FOLDER_TO_SAVE, grid_size=(3, 3), limit_size_layer=(15, 15), PLOT_MODEL=True): """ :param model: keras :type model: Sequential :param pattern: , :type pattern: np.array :param FOLDER_TO_SAVE: :type FOLDER_TO_SAVE: str :param grid_size: :type grid_size: tuple :param limit_size_layer: :type limit_size_layer: tuple :param PLOT_MODEL: :type PLOT_MODEL: PLOT_MODEL """ SAVE_AR_LIST = [] for num_layer in range(1, len(model.layers)): LO = model.layers[num_layer].output _model = Model(inputs=model.input, outputs=LO) if ( len(_model.output_shape) == 3 and _model.output_shape[1] > limit_size_layer[0] and _model.output_shape[2] > limit_size_layer[1] ): _output = _model.predict(pattern)[0] SAVE_AR_LIST.append( [ num_layer, model.layers[num_layer].name, _output.tolist() ] )
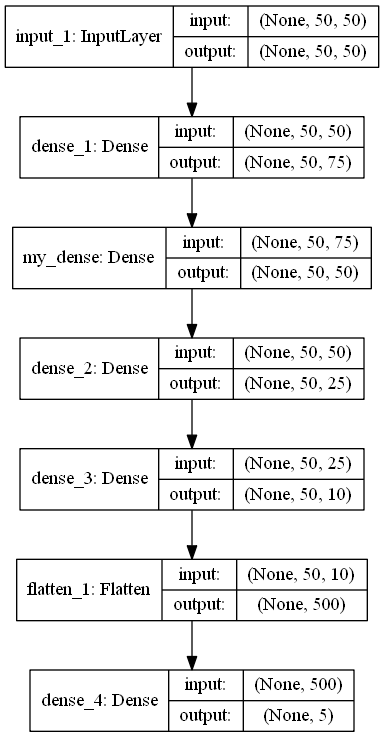
def build_model (IN_SHAPE = 50, CLASSES = 5) -> Sequencial: def build_model(IN_SHAPE=50,CLASSES=5) -> Sequential: inputs_LAYER0 = Input(shape=(IN_SHAPE,IN_SHAPE)) Dense_2_2 = Dense(75, activation='relu')(inputs_LAYER0) Dense_2_3 = Dense(50, activation='relu', name="my_dense")(Dense_2_2) Dense_2_4 = Dense(25, activation='relu')(Dense_2_3) Dense_2_5 = Dense(10, activation='relu')(Dense_2_4) flat_f_0 = Flatten()(Dense_2_5) final_layer= Dense(CLASSES, activation='softmax')(flat_f_0)
Código do programa
model_ = build_model() pattern = np.random.sample((1,50,50)) os.makedirs("PLOT_PATTERN_PROCCESS") PLOT_PATTERN_PROCCESS( model = model_, pattern = pattern, FOLDER_TO_SAVE = "PLOT_PATTERN_PROCCESS", PLOT_MODEL=False, grid_size=(2, 2) )
Descrição do Programa
Método
build_model()
retorna um modelo ANN no formato
Sequencial , projetado para classificar algo em 5 classes.
model.summary () _________________________________________________________________ Layer (type) Output Shape Param
Como você pode ver na arquitetura, um padrão é uma matriz de tamanho 50x50. Variável
pattern
e há um objeto observável.
Em seguida, um diretório é criado
os.makedirs("PLOT_PATTERN_PROCCESS")
,
onde todo o resultado será salvo.
Método Descrição PLOT_PATTERN_PROCCESS
Descrevi o significado do método acima, mas é importante dizer que não precisamos de todas as camadas, pois as saídas de algumas camadas não podem ser exibidas ou isso não será informativo.
A obtenção do padrão de saída ocorre aqui:
_output = _model.predict(pattern)[0]
Nesta implementação, você pode exibir um padrão de saída bidimensional cujas dimensões não são menores que o parâmetro
limit_size_layer
Alternando através das camadas do modelo RNA, a variável
SAVE_AR_LIST
gradualmente preenchido com dados:
- Número da camada
num_layer
- Nome da camada
model.layers[num_layer].name
- Matriz bidimensional de saída
_output.tolist()
Excluir gradualmente um resultado de
SAVE_AR_LIST
,
e colocá-lo na célula da tela
ax.imshow(np.array(ar), cmap='viridis', extent=(xmin, xmax, ymin, ymax))
.
O resultado é um arquivo
(0.png)
Recomendações
Boa sorte