 Protocolo experimental e implementação de um algoritmo de classificação em um computador de DNA programável
Protocolo experimental e implementação de um algoritmo de classificação em um computador de DNA programávelOs cientistas há muito experimentam armazenar informações no DNA e processar essas informações. Por exemplo, cientistas da Universidade de Washington e da Microsoft construíram recentemente o
"primeiro DNA Winchester do mundo" (
foto ). Esse projeto é capaz pela primeira vez de fornecer o registro e a leitura de informações no armazenamento de DNA sem intervenção humana. Uma conquista muito significativa, pois o DNA pode registrar informações com uma densidade de
2,2 petabytes por grama . O DNA é um recipiente compacto com uma densidade de gravação milhares de vezes maior que os portadores existentes.
No entanto, todos os sistemas de DNA existentes têm um problema: todos esses são desenvolvimentos proprietários exclusivos que carecem de flexibilidade. Se compararmos com a tecnologia de silício, cada grupo de pesquisadores desenvolve uma nova arquitetura de computadores, para a qual você precisa escrever um novo software. Mas as coisas podem mudar graças ao
primeiro computador de DNA programável desenvolvido na Universidade da Califórnia, em Davis (UC Davis), no Instituto de Tecnologia da Califórnia e na Universidade de Maynooth.
O primeiro computador programável de DNA é descrito em um
artigo científico publicado em 20 de março de 2019 na revista Nature. Os autores mostraram que, com a ajuda de um simples gatilho, o mesmo conjunto básico de moléculas de DNA é capaz de implementar muitos algoritmos diferentes. Embora o estudo seja um experimento puramente de laboratório, algoritmos moleculares programáveis no futuro podem ser usados, por exemplo, para programar robôs de DNA que já
entregam medicamentos com sucesso
a células cancerígenas .
"Este é um dos trabalhos de referência nesta área",
diz Torsten-Lars Schmidt, professor assistente de biofísica experimental da Universidade de Kent, que não participou da pesquisa. "Eles costumavam demonstrar automontagem algorítmica, mas não com esse grau de complexidade".
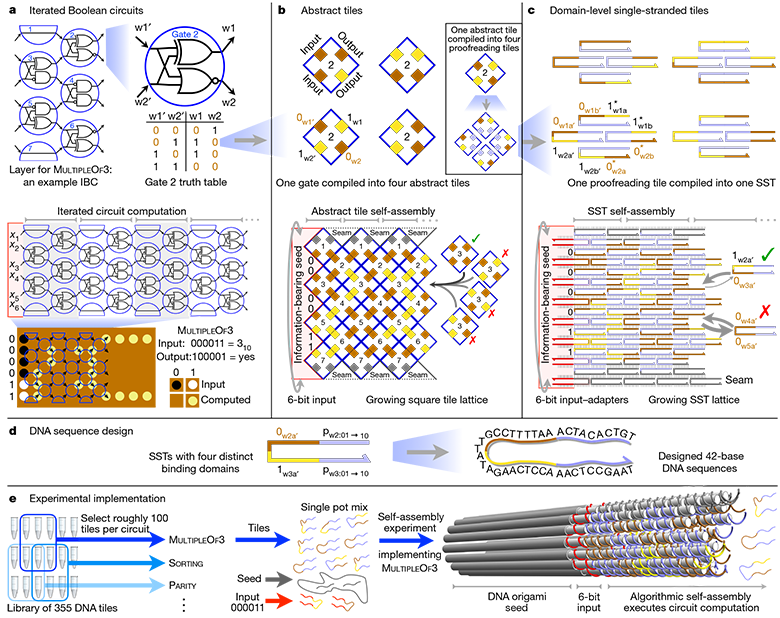
Nos computadores eletrônicos, os bits são unidades binárias de informação. Eles representam o estado físico discreto do equipamento básico, por exemplo, a presença ou ausência de corrente elétrica. Esses bits, ou melhor, sinais elétricos, são passados através de circuitos que consistem em elementos lógicos que executam uma operação em um ou mais bits de entrada e produzem um bit como saída.
Combinando esses blocos de construção simples repetidamente, os computadores podem executar programas incrivelmente complexos. A idéia da computação em DNA é substituir sinais elétricos por ligações químicas e silício por ácidos nucleicos para criar software biomolecular.
 Hierarquia abstrata da arquitetura e implementação prática do circuito lógico IBC de 6 bits completo (Circuito Booleano Iterado)
Hierarquia abstrata da arquitetura e implementação prática do circuito lógico IBC de 6 bits completo (Circuito Booleano Iterado)De acordo com Eric Winfrey, cientista do Instituto de Tecnologia da Califórnia e coautor do artigo, os algoritmos moleculares usam as possibilidades naturais de processar informações no DNA, mas, em vez de permitir que a natureza tome as rédeas com suas próprias mãos, os cálculos no DNA são feitos de acordo com um programa escrito pelo homem.
Nos últimos 20 anos, várias experiências bem-sucedidas com algoritmos moleculares foram realizadas, por exemplo, para jogar tic-tac-toe ou montar moléculas de várias formas. Em cada caso, foi necessário um cuidadoso desenvolvimento da sequência de DNA para executar um algoritmo específico que geraria a estrutura do DNA. Nesse caso, a diferença é que os pesquisadores desenvolveram um sistema no qual os mesmos fragmentos básicos de DNA podem ser ordenados para criar
algoritmos completamente diferentes - e, portanto, para obter resultados completamente diferentes.
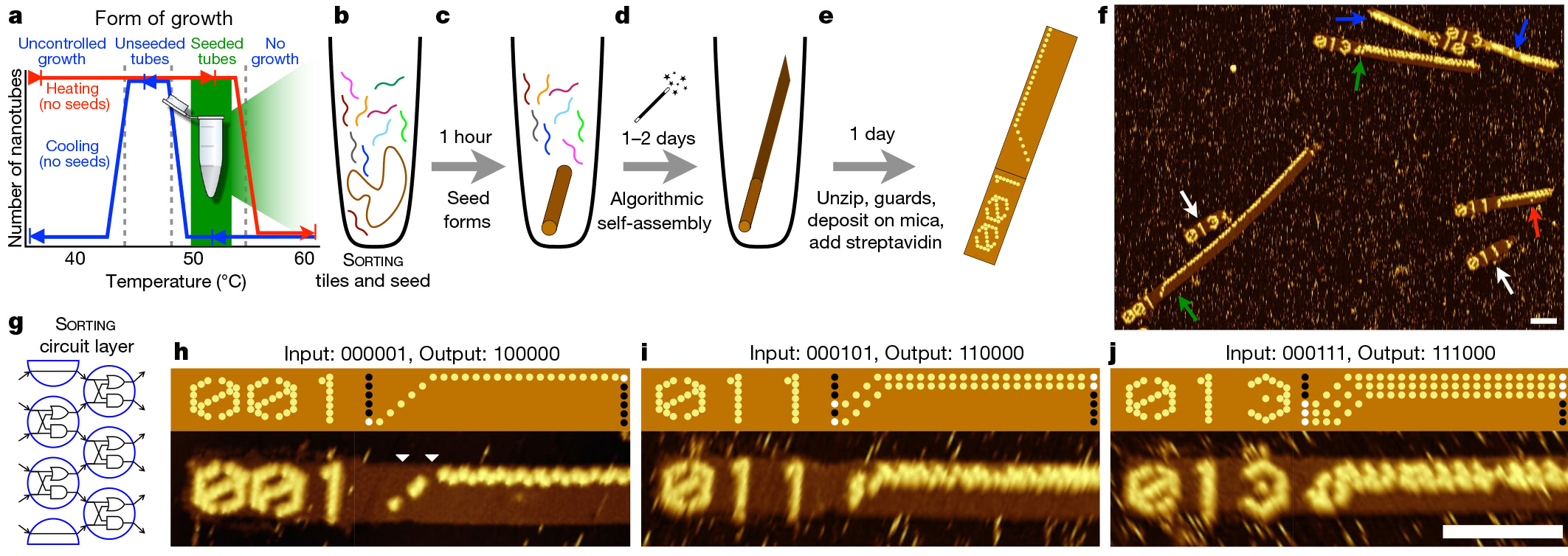
O processo começa com a técnica de origami de DNA, ou seja, dobrar uma longa cadeia de DNA na forma desejada. Essa fatia dobrada funciona como uma semente, que executa uma linha de montagem algorítmica. A semente permanece praticamente inalterada, independentemente do algoritmo. Para cada experimento, apenas pequenas alterações são feitas em várias seqüências.
 Reprogramando o circuito lógico
Reprogramando o circuito lógicoApós criar a "semente", ela é adicionada à solução com centenas de outras cadeias de DNA, conhecidas como blocos de DNA. Os cientistas desenvolveram 355 dessas peças. Cada um tem um arranjo único de bases nitrogenadas. Assim, para cada algoritmo, os pesquisadores simplesmente escolhem um conjunto diferente de blocos iniciais. Como esses fragmentos de DNA são unidos durante o processo de montagem, eles formam um circuito que implementa o algoritmo molecular selecionado nos bits de entrada fornecidos pela "semente".
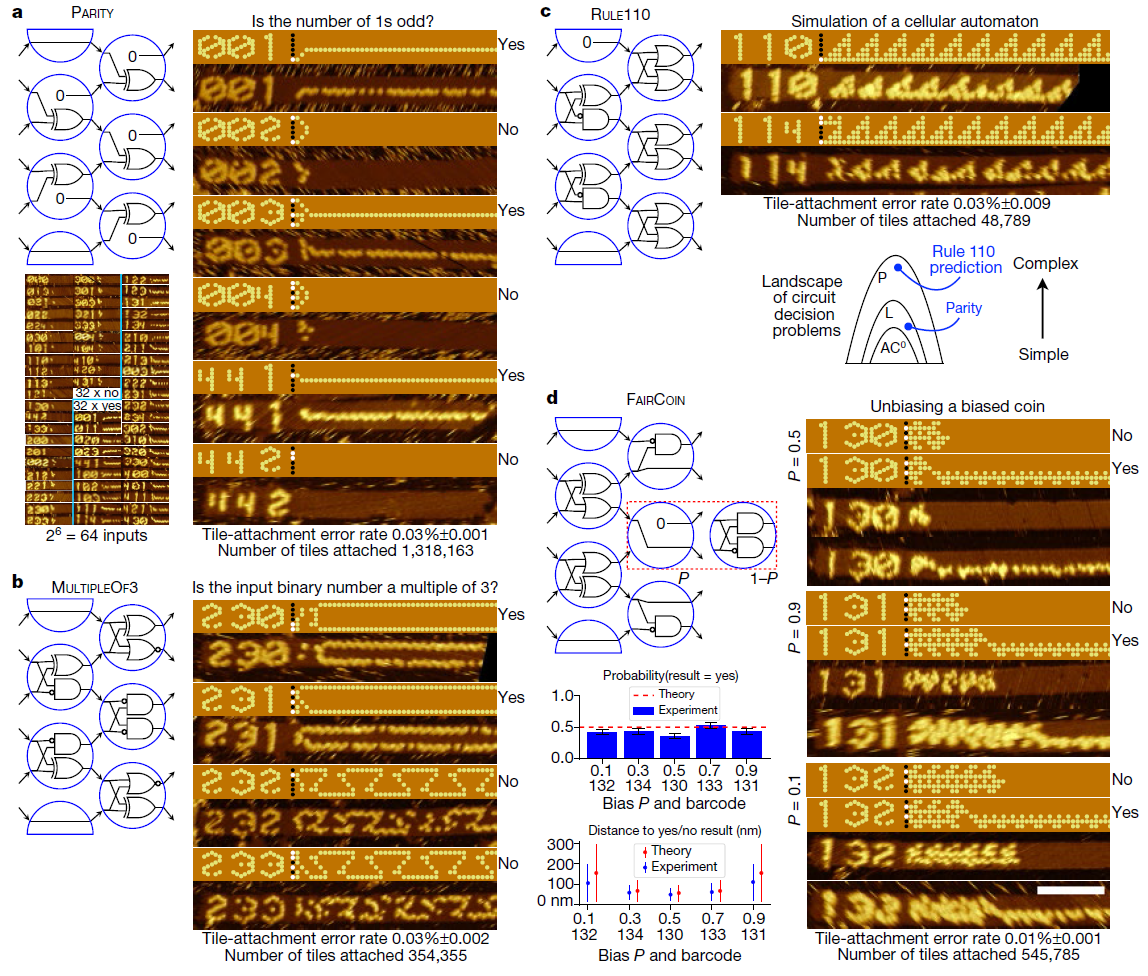
Usando esse sistema, os pesquisadores desenvolveram e testaram 21 algoritmos para executar tarefas como reconhecer uma divisão por três,
escolher um líder , gerar padrões e contar de 0 a 63. Todos esses algoritmos são implementados usando diferentes combinações dos mesmos 355 blocos de DNA.
Obviamente, não é fácil escrever código colocando fragmentos de DNA em um tubo de ensaio, mas se o processo é automatizado, os futuros programadores moleculares nem precisam pensar em biomecânica, pois os programadores de hoje não precisam entender a física dos transistores para escrever bons programas.