Este artigo discute um caso para acelerar um aplicativo de navegador substituindo JavaScript por WebAssembly.
WebAssembly - o que é isso?
Em resumo, este é um formato de instrução binário para uma máquina virtual empilhada. Wasm (nome abreviado) é freqüentemente chamado de linguagem de programação, mas não é. O formato da instrução é executado no navegador junto com o JavaScript.
É importante que o WebAssembly possa ser obtido compilando fontes em idiomas como C / C ++, Rust, Go. Ele usa digitação estática e o chamado modelo de memória plana. O código, como mencionado acima, é armazenado em um formato binário compacto, portanto, é executado quase tão rápido como se o aplicativo fosse iniciado usando a linha de comando. Esses recursos levaram à crescente popularidade do WebAssembly.
Lembramos que: para todos os leitores de "Habr" - um desconto de 10.000 rublos ao se inscrever em qualquer curso Skillbox usando o código promocional "Habr".
A Skillbox recomenda: Curso prático "Mobile Developer PRO" .
Atualmente, o Wasm é usado em muitos aplicativos, de jogos como Doom 3 a portados a aplicativos da Web como Autocad e Figma. Wasm também é usado em áreas como computação sem servidor.
Este artigo fornece um exemplo do uso do Wasm para acelerar um serviço da web analítico. Para maior clareza, adotamos um aplicativo de trabalho escrito em C, que é compilado no WebAssembly. O resultado será usado para substituir as seções de baixa produtividade do JS.
Transformação de aplicativo
O exemplo usará o serviço de navegador fastq.bio, destinado a geneticistas. A ferramenta permite avaliar a qualidade do seqüenciamento de DNA (decodificação).
Aqui está um exemplo de aplicativo em trabalho:

Os detalhes do processo não devem ser fornecidos, pois são bastante complicados para não especialistas, mas, em resumo, os cientistas das infográficos acima podem entender se o processo de seqüenciamento de DNA ocorreu sem problemas e que problemas surgiram.
Este serviço tem alternativas, programas de desktop. Mas o fastq.bio pode acelerar as coisas visualizando dados. Na maioria dos outros casos, você precisa trabalhar com a linha de comando, mas nem todos os geneticistas têm a experiência necessária.
Tudo funciona simplesmente. Na entrada - dados apresentados na forma de um arquivo de texto. Este arquivo é gerado por ferramentas especializadas de seqüenciamento. O arquivo contém uma lista de sequências de DNA e uma avaliação de qualidade para cada nucleotídeo. O formato do arquivo é .fastq, e é por isso que o serviço recebeu esse nome.
Implementação de JavaScript
A primeira etapa do usuário ao trabalhar com fastq.bio é selecionar o arquivo apropriado. Usando o objeto File, o aplicativo lê uma seleção aleatória de dados do arquivo e processa este pacote. A tarefa do JavaScript aqui é executar operações simples de string e contar indicadores. Um deles é o número de nucleotídeos A, C, G e T em diferentes fragmentos de DNA.
Após o cálculo dos indicadores necessários, eles são visualizados usando o Plotly.js, e o serviço começa a trabalhar com uma nova seleção de dados. A fragmentação é feita para melhorar a qualidade do UX. Se você trabalhar com todos os dados de uma só vez, o processo será congelado por um tempo, pois os arquivos com resultados de seqüenciamento ocupam centenas de gigabytes de espaço no arquivo. O serviço pega pedaços de dados com tamanho de 0,5 a 1 MB e trabalha com eles passo a passo, criando dados gráficos.
Veja como funciona:

O retângulo vermelho contém o algoritmo de conversão de string para renderização. Essa é a parte mais carregada do serviço em termos de computação. Vale a pena tentar substituí-lo por Wasm.
Testando o WebAssembly
Para avaliar a possibilidade de usar o Wasm, a equipe do projeto começou a procurar soluções prontas para criar métricas de CQ (CQ - controle de qualidade) com base em arquivos fastq. A pesquisa foi realizada entre as ferramentas escritas em C, C ++ ou Rust, para que fosse possível portar o código para o WebAssembly. Além disso, a ferramenta não deve ser "bruta", requer um serviço já verificado pelos cientistas.
Como resultado, a escolha foi feita em favor do
seqtk . O aplicativo é bastante popular, é de código aberto, o idioma de origem é C.
Antes de converter para Wasm, observe o princípio de compilação do seqtk para a área de trabalho. De acordo com o Makefile, eis o que você precisa:
# Compile to binary $ gcc seqtk.c \ -o seqtk \ -O2 \ -lm \ -lz
Basicamente, você pode compilar o seqtk com o Emscripten. Se não estiver lá, contorne
a imagem do Docker .
$ docker pull robertaboukhalil/emsdk:1.38.26 $ docker run -dt --name wasm-seqtk robertaboukhalil/emsdk:1.38.26
Se desejar,
você pode coletar você mesmo , mas isso leva tempo.
Dentro do contêiner, você pode facilmente usar o emcc como uma alternativa ao gcc:
# Compile to WebAssembly $ emcc seqtk.c \ -o seqtk.js \ -O2 \ -lm \ -s USE_ZLIB=1 \ -s FORCE_FILESYSTEM=1
Mudanças mínimas:
Em vez de enviar para o arquivo binário Emscripten, .wasm e .js são usados para gerar os arquivos, que são usados para executar o módulo WebAssemby.
Para suportar a biblioteca zlib, o sinalizador USE_ZLIB é usado. A biblioteca é distribuída e portada para o WebAssembly e o Emscripten a inclui no projeto.
O sistema de arquivos virtual do Emscrippten está ativado. Este é um
FS do tipo POSIX operando na RAM dentro do navegador. Quando a página é atualizada, a memória é limpa.
Para entender por que um sistema de arquivos virtual é necessário, vale a pena comparar a maneira de iniciar o seqtk na linha de comando com a maneira de executar o módulo WebAssembly compilado.
# On the command line $ ./seqtk fqchk data.fastq # In the browser console > Module.callMain(["fqchk", "data.fastq"])
É necessário obter acesso ao sistema de arquivos virtual para não sobrescrever seqtk por string em vez de entrada de arquivo. Nesse caso, o fragmento de dados é exibido como o arquivo data.fastq no FS virtual com o main () seqtk chamado.
Aqui está a nova arquitetura:
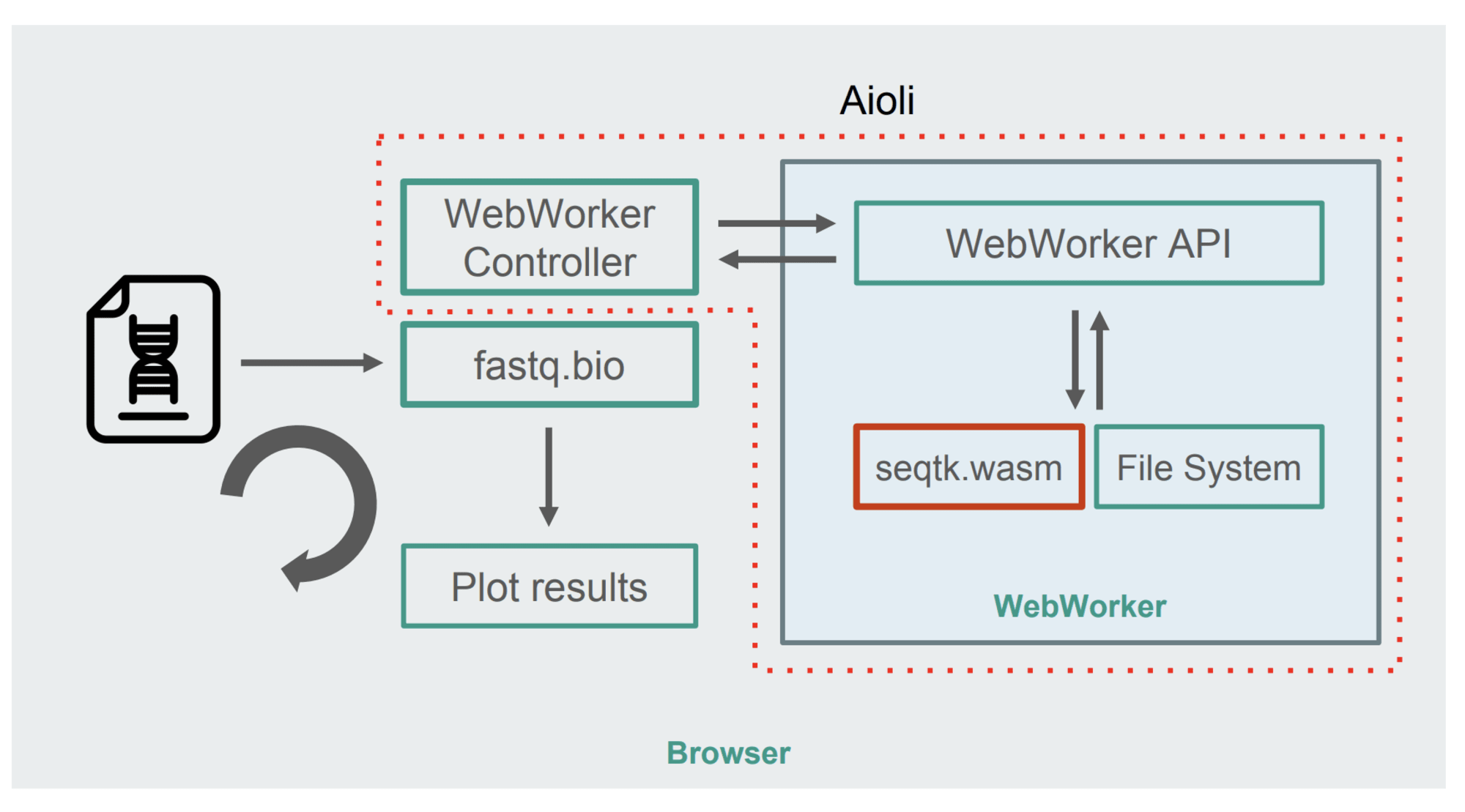
A figura mostra que, em vez de computar, o fluxo principal do navegador usa
WebWorkers . Esse método permite realizar cálculos no encadeamento em segundo plano sem afetar a capacidade de resposta do navegador. Bem, o controlador WebWorker inicia o Worker, controlando sua interação com o thread principal.
O comando seqtk é iniciado usando o Worker no arquivo montado. Após a conclusão do trabalho, o Trabalhador exibe o resultado na forma de Promessa. Quando uma mensagem é recebida pelo encadeamento principal, o resultado é usado para atualizar os agendamentos. E assim, em algumas iterações.
E o desempenho do WebAssembly?
Para avaliar a mudança no desempenho, a equipe do projeto usou o parâmetro do número de operações de leitura por segundo. O tempo do gráfico interativo não é levado em consideração, porque as duas implementações usam JavaScript.
Ao usar a solução pronta para uso, o ganho de desempenho foi nove vezes.

Este é um excelente resultado, mas, como se viu, há uma oportunidade de otimizá-lo. O fato é que um grande número de resultados da análise de CQ não é usado pelo seqtk, portanto, você pode excluí-los. Se você fizer isso, o resultado comparado ao JS será aprimorado 13 vezes.
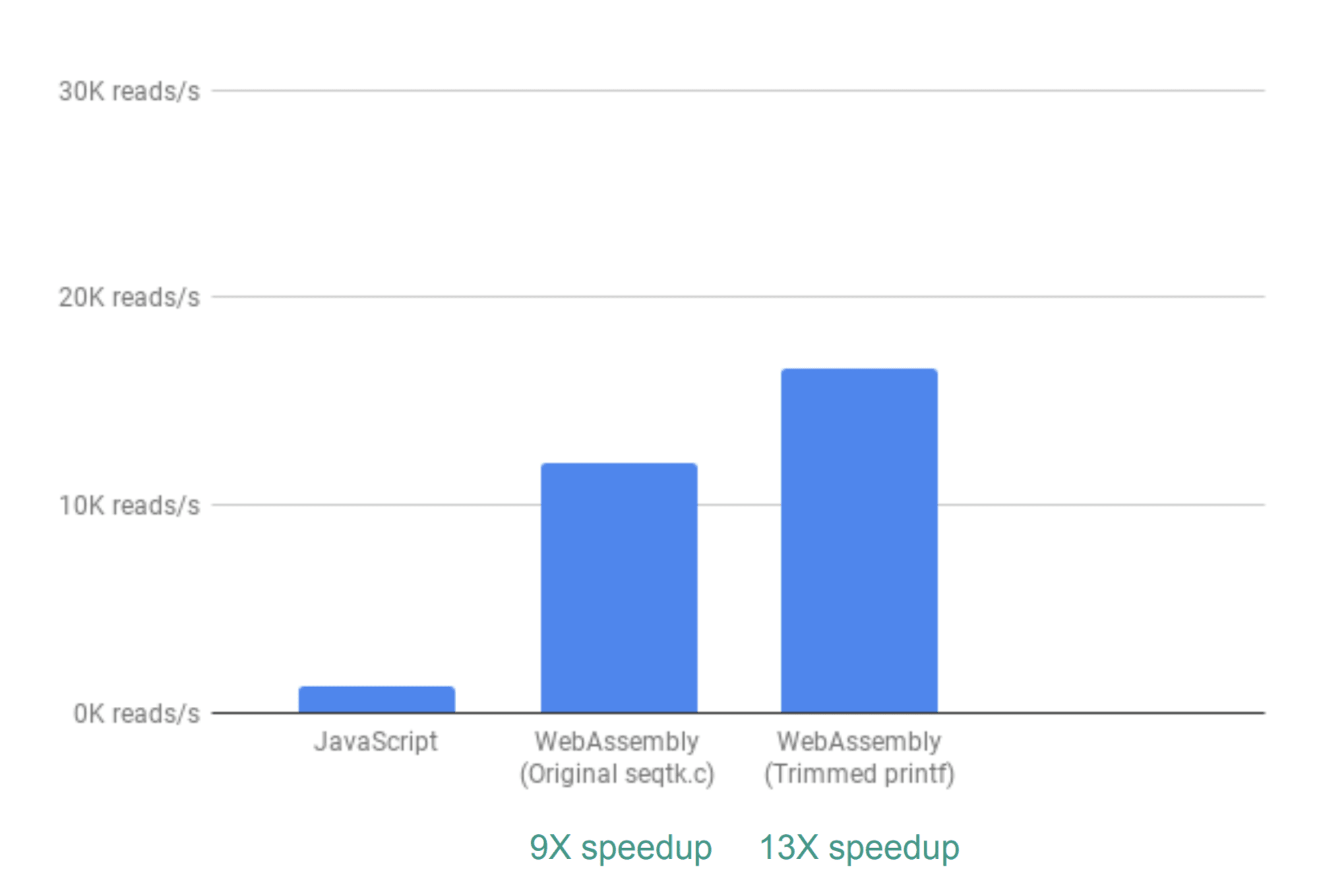
Foi possível alcançá-lo simplesmente comentando os comandos printf ().
Mas isso não é tudo. O fato é que, neste estágio, o fastq.bio recebe os resultados da análise chamando funções diferentes de C. Qualquer uma calcula seu próprio conjunto de características, de modo que cada fragmento do arquivo seja lido duas vezes.
Para contornar esse problema, foi decidido combinar as duas funções em uma. Como resultado, a produtividade aumentou 20 vezes.

Vale ressaltar que nem sempre esse resultado extraordinário pode ser alcançado. Em alguns casos, o desempenho diminui, portanto vale a pena avaliar cada caso.
Como conclusão, podemos dizer que o Wasm realmente permite melhorar o desempenho do aplicativo, mas você precisa usá-lo com sabedoria.
A Skillbox recomenda: