Antes de ler esta atualização, é aconselhável se familiarizar com o código fonte publicado anteriormente .O primeiro e mais popular seqüenciador de poros de nanion MinION (o conjunto mínimo no Reino Unido é de US $ 1000, na Rússia - 150 mil rublos), desenvolvido pela Oxford Nanopore Technologies (ONT), opera em células descartáveis, cada uma custando US $ 900 (em Rússia - 135 mil rublos). Essas células permitem digitalizar o DNA 10 ... 20 Gb em 2 ... 3 dias. Isso é muito pouco para sequenciar o genoma humano (você precisa de> 100 Gb), mas é demais para todos os outros fins de diagnóstico clínico (<1 Gb é suficiente).
Recentemente, começou a vender modelos mais produtivos (e mais caros) - PromethION 24/48 (US $ 165.000 / US $ 285.000), voltado principalmente para o seqüenciamento em linha do genoma completo. Mais caros são os consumíveis mais eficientes (US $ 2.000 / unidade,> 100 Gb). É verdade que eles podem custar a granel mais barato (US $ 1.800.000 por 2.880 peças, US $ 625 / peças). Mas essas aquisições (200 a 250 milhões de rublos por lote) não são permitidas no orçamento russo.
E não apenas para o russo. Portanto, a atenção dos visitantes da recente conferência da ONT (22 a 24 de maio) foi atraída pelo anúncio do início das vendas do Flongle, um adaptador para o sequenciador MinION, que permite trabalhar com células descartáveis menos eficientes (~ 1 Gb), mas relativamente baratas (US $ 90).
 https://nanoporetech.com/products
https://nanoporetech.com/productsO aparecimento de Flongle pode causar um crescimento explosivo no uso do NGS (sequenciamento de próxima geração) na prática clínica e substituir os diagnósticos de PCR convencionais, cujo tamanho de mercado na Rússia excede um bilhão de rublos e no mundo é medido em bilhões de dólares. Mas para a maioria dos pacientes russos, o diagnóstico de NGS continuará sendo um luxo inadmissível, já que o custo das células de Flongle no mercado doméstico será superior a 10 mil rublos. E se somarmos a isso o custo dos reagentes descartáveis, os custos de transporte e as despesas gerais, o custo estimado do sequenciamento de uma amostra de DNA / RNA não cairá para menos de 20 mil rublos.
Isso implica a necessidade de desenvolver seqüenciadores de nanoporos domésticos e fornecer a eles consumíveis e reagentes baratos.
Nanorider
É possível reduzir significativamente o custo do seqüenciamento de nanoporos, reutilizando células para Flongle e importando a substituição dos reagentes necessários para trabalhar com eles. Mas primeiro você precisa de um dispositivo de teste que permita monitorar a formação de membranas lipídicas de bicamada (BLM) em 126 poços da célula em funcionamento e avaliar a quantidade e a qualidade dos nanoporos incorporados nessas membranas. Esse dispositivo ("Nanorider"?) Deve rastrear alterações na condutividade elétrica (e / ou impedância) de orifícios individuais durante a formação de BLMs neles e registrar correntes picoamperosas que fluem através de canais de íons únicos.
É possível compor um leitor conectado ao computador via USB 2.0 a partir de elementos padrão: FPGA (100 ... 200 $), amplificadores de corrente (MAX9923FEUB - 200 ... 300 rublos), ADC (1 ... 2 mil rublos), etc. apenas uma placa com um pad de contato não padrão (10x13) tipo LGA (Land Grid Array) para acoplar a uma célula da Flongle. Ou com sua contraparte doméstica simplificada contendo um número menor (16 ... 32) de poços sensores.
 Touch Pad Cell Flongle
Touch Pad Cell FlongleReduzir a produtividade celular calculada em 4 ... 8 vezes (até 10 ... 20 Mb) não é crítico para resolver muitos problemas (determinar patógenos, tipagem HLA, estabelecer paternidade, identificar uma pessoa etc.), mas permite montar um seqüenciador de nanoporos usando amplificadores convencionais correntes de picoampere. E sem o uso de chips especiais de 512 canais, cujo desenvolvimento os Oxfordians investiram dezenas de milhões de dólares. É verdade que cada nanoreader multicanal deve conter mais de uma dúzia de amplificadores de corrente + ADCs, mas ainda pode custar várias vezes menos que a combinação proprietária MinION + Flongle (> 200 mil rublos). Quanto ao baixo desempenho do nanoreader, para usuários russos que não possuem computadores decentes (médicos, biólogos, biohackers etc.), ele pode até ser útil.
Kilorider
Os amplificadores de corrente picoampere de 512 canais desenvolvidos pelos Oxfordians estão contidos em cada célula descartável para o MinION. As células residuais podem ser descartadas, mas é melhor usar os chips contidos nelas para produzir análogos de Flongle que são caracterizados pelo aumento da produtividade. Se esses chips estiverem equipados com contatos de superfície, eles poderão trabalhar com células de contato contendo poços sensores de 2048 (512x4).
Projetar células com esses LGAs não será fácil, mas é necessário levar em consideração que os requisitos de contato aqui são menos rigorosos que os processadores e placas-mãe, nos quais um contato ruim pode desativar todo o sistema. Para um seqüenciador, a inoperabilidade de até metade dos contatos da célula pode ser considerada aceitável. A metade restante é suficiente para garantir o desempenho no nível de 5 ... 10 Gb.
O desenvolvimento lógico dessa idéia será a combinação de vários leitores semelhantes em um dispositivo. Isso permitirá que você obtenha um seqüenciador analógico GridION X5, projetado para trabalho simultâneo com cinco (ou mais) células reutilizáveis da produção doméstica. Esse seqüenciador pode estar em demanda em muitos laboratórios de diagnóstico clínico.
 https://store.nanoporetech.com/devices
https://store.nanoporetech.com/devicesAs células seqüenciadoras PromethION descartáveis contêm 9.000 poços sensores nos chips. E os próprios chips são amplificadores de 3000 canais de correntes de picoampere. O fluxo de informações que eles fornecem com 24 (com o PromethION 24) ou 48 (com o PromethION 48) é tão grande que, na forma "bruta", não pode ser transferido para um computador externo comum. Portanto, seqüenciadores desse tipo devem estar equipados com seu próprio supercomputador. Mas se o seqüenciador funcionar apenas com uma célula, ele poderá ser conectado a um laptop comum para jogos com uma porta Thunderbolt 3.0.
A alteração de chips livres (ejetados) do ONT para trabalhar com células de contato não é um problema insolúvel. Mas os seqüenciadores construídos com o uso de tais chips só podem funcionar com o software ONT. E os esforços que serão necessários para refazer esses programas podem negar os benefícios do uso de chips disponíveis no mercado. E desenvolver esses chips do zero é muito caro.
Mega Rider
Este ano, o teste beta do seqüenciador de nanoporos da AXBIO (EUA) deve começar. Ele usa chips com um milhão de canais de amplificação de corrente produzidos no Japão pela empresa israelense Tower Semiconductor Ltd ... Consumíveis e a venda desses sequenciadores serão gerenciados pela filial chinesa da AXBIO.

Nos Estados Unidos e na União Européia, mais de 7.000 patentes são dedicadas ao seqüenciamento de nanoporos, o que cria riscos financeiros imprevisíveis para as empresas locais que tentam lidar com essas tecnologias. Aparentemente, esta é a principal razão para uma internacionalização tão alta desse desenvolvimento. E permite que empresas e laboratórios russos participem, sem restrições, em testes e aprimoramentos da tecnologia AXBIO, além de produzir (ou coletar) esses seqüenciadores em seu território.
Terabytes de dados brutos obtidos por um seqüenciador de nanoporos com chips megapixel (mega leitor), é desejável filtrar. E transfira para o computador apenas as leituras mais longas e da mais alta qualidade. Isso não apenas melhora a qualidade do seqüenciamento, mas também reduz os requisitos para o computador, o que também é importante para a produção em massa e o uso generalizado de sequenciadores genômicos. Além disso, a produtividade excessiva dos mega leitores pode reduzir a duração do seqüenciamento do genoma humano de 2 ... 3 dias (para ONT) para várias horas.
Soma da tecnologia
O sucesso da ONT foi facilitado pela organização da otimização de tubulação paralela dos cinco principais elementos da tecnologia de seqüenciamento de nanoporos - membranas, nanoporos, “motor” (helicase), condições de sequenciamento e algoritmos de decodificação para sinais de leitura.
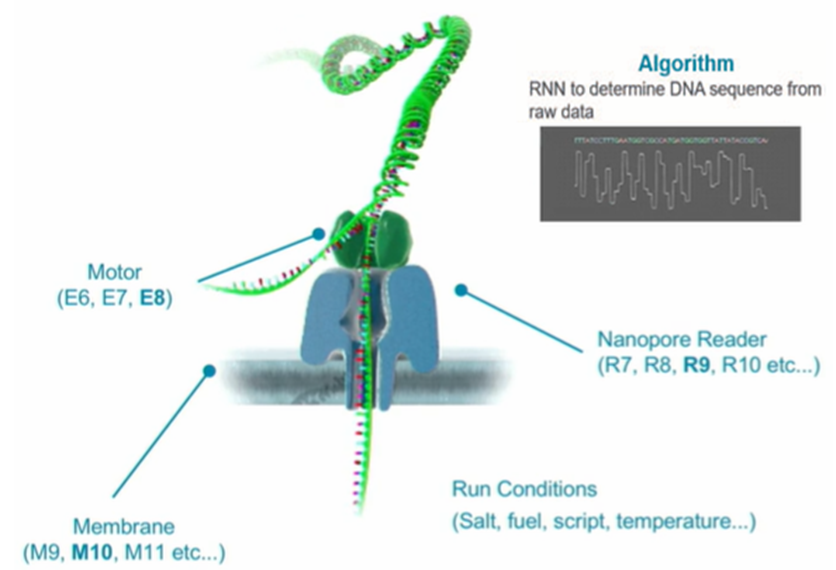 https://nanoporetech.com/resource-centre/videos/sub1000
https://nanoporetech.com/resource-centre/videos/sub1000O prazo de validade garantido das células para o seqüenciamento de nanoporos depende principalmente da estabilidade da membrana lipídica da bicamada. Agora são 6 semanas, embora geralmente essas membranas sejam destruídas em poucos dias. Para aumentar sua vida útil, são utilizados aditivos poliméricos hidrofóbicos, copolímeros de blocos ativos de superfície, colesterol, antioxidantes, reticulação de ácidos graxos, etc., e essas modificações podem interferir na formação de canais iônicos. Portanto, é muito difícil alcançar a estabilidade do BLM.
A resolução radical desse problema permite a formação de membranas nas células de trabalho do seqüenciador e a introdução de nanoporos nelas imediatamente antes do uso. A auto-preparação de células para o trabalho fornecerá a oportunidade para sua regeneração e reutilização. Quanto ao prazo de validade, para células secas é quase ilimitado, e os reagentes necessários para sua ativação e regeneração podem ser armazenados por meses.
O gargalo na tecnologia do seqüenciamento de nanofios é ao mesmo tempo o gargalo dos nanoporos, do qual depende a qualidade das informações lidas. Portanto, uma das principais tarefas do ONT foi a busca por uma proteína de membrana, que permita obter resultados de qualidade aceitável.
A primeira dessas proteínas foi a CsgG, uma proteína da membrana externa encontrada em muitas bactérias gram-negativas. Nove de suas subunidades formam um canal de íons na membrana, com amplos vestíbulos de entrada e saída e um gargalo. Mas foi possível obter nanoporos de alta qualidade suficiente (R9, R9.4.1, R9.5.1) somente após enumerar e analisar centenas de variantes CsgG modificadas. Neste verão, as células com poros da próxima geração (R10 ou R10b) devem aparecer à venda, apresentando maior precisão na leitura de repetições de homopolímeros.
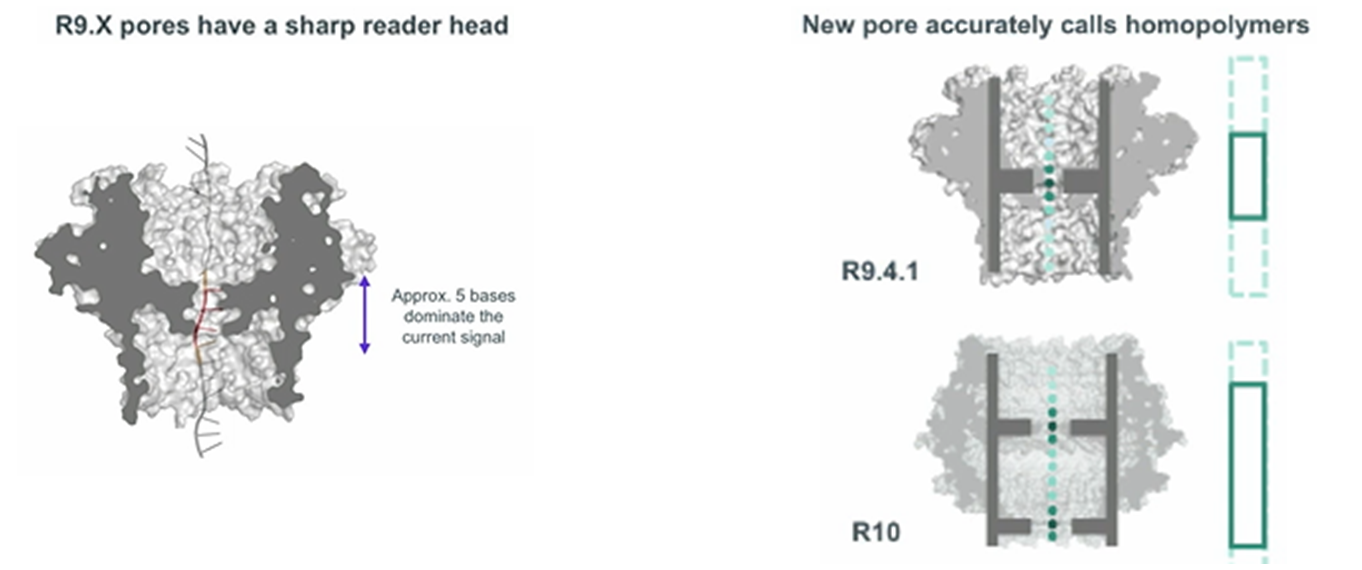 https://nanoporetech.com/about-us/news/london-calling-clive-brown-and-team-plenary
https://nanoporetech.com/about-us/news/london-calling-clive-brown-and-team-plenaryO ONT é um componente obrigatório da tecnologia de sequenciamento de nanoporos da ONT, uma helicase, um motor molecular que desenrola o DNA de fita dupla e inibe o progresso do DNA de fita única através de um nanoporo. A velocidade máxima do desenrolamento do DNA por esse motor agora atinge 450 pares de nucleotídeos por segundo, embora a velocidade dos componentes eletrônicos do MinION permita que ele seja aumentado para 1000 nucleotídeos por segundo. Teoricamente, acelerar o trabalho do helicase pode dobrar o desempenho do MinION, mas na prática isso pode levar a um sequenciamento ruim. Portanto, é improvável que helicases mais rápidas e eficientes melhorem o desempenho de seqüenciadores de nanoporos, como MinION e PromethION.
A qualidade do seqüenciamento depende de muitas condições - a magnitude da tensão aplicada à membrana, a condutividade elétrica e a composição iônica da mistura de reação, a quantidade e a natureza das impurezas que entopem os poros, a capacidade de limpar os poros entupidos, alterando a polaridade do potencial, a estrutura dos adaptadores utilizados, etc., etc. o sequenciamento requer otimização cuidadosa de todos os seus parâmetros e composições de soluções de trabalho (condições de execução), o que pode afetar adversamente a qualidade da digitalização das sequências de nucleotídeos awns. Por exemplo, recentemente foi demonstrado que uma diminuição na concentração de ATP clivada pela helicase diminui gradualmente a precisão dos resultados do seqüenciamento, e agora os Oxfordians estão tentando eliminar esse problema introduzindo um sistema de regeneração de ATP na mistura de reação.
O quinto elemento da tecnologia de seqüenciamento de nanoporos é a inteligência artificial construída em redes neurais recorrentes (RNN) e o uso de alguns algoritmos e programas especiais que melhoram o reconhecimento de repetições homogêneas (Flip-flop, Medaka). Em julho, a próxima versão dos programas Guppi e MinKNOW deve aparecer, na qual os Oxfordians prometem adicionar a capacidade de determinar bases metiladas (5mC e 6mA). Isso é importante não apenas para estudos epigenômicos, mas também para melhorar o reconhecimento de bases convencionais.
O grande volume de informações lidas pelos seqüenciadores de nanoporos e a complexidade de seu processamento impõem requisitos maiores ao hardware de tais softwares e sistemas de hardware. Portanto, nem todo computador é adequado para conectar o MinION, a última geração de aceleradores gráficos é instalada no GridION e no PromethION, e a Oxford Nanopore Technologies se tornou um dos primeiros compradores de um grande lote de módulos Jetson AGX Xavier (NVIDIA) destinados a dispositivos com inteligência artificial.
Mais recentemente, a tecnologia nanopore foi considerada nada mais que um complemento útil para tecnologias mais precisas de fluorescência e seqüenciamento de semicondutores. Mas um aumento gradual na precisão da leitura do DNA, combinado com um grande número de seqüências legíveis, permitiu que, se não fosse à frente de todos os concorrentes, determinasse pelo menos o principal vetor de desenvolvimento das tecnologias de sequenciamento genômico.

Pode demorar um pouco para dominar todos os elementos da tecnologia de seqüenciamento de nanoporos, mas esse não é o maior problema. O principal problema é a falta de financiamento direcionado para tais desenvolvimentos integrados na Rússia. É verdade que o recém-aprovado Programa Científico e Técnico Federal para o Desenvolvimento de Tecnologias Genéticas para 2019-2027 (seção "Instruções de implementação do programa") diz que, a curto prazo (3 a 6 anos), será desenvolvido um protótipo de sequenciamento genômico de alto desempenho. Mas encontrar essa única linha em 30 páginas de texto não é tão fácil. Mais fácil negociar com os chineses na compra de sequenciadores AXBIO.
Mais simples ainda é encontrar artesãos capazes de montar o nanorider mais simples "no joelho". E então ande com a mão estendida em busca de investidores que concordem em financiar o desenvolvimento da tecnologia doméstica de seqüenciamento de nanoporos.