Esta é a segunda parte do artigo sobre numba. A primeira foi uma introdução histórica e um breve manual de instruções numba. Apresento aqui um código de tarefa ligeiramente modificado do artigo Haskell “ Faster than C ++; mais lento que o PHP ”(compara o desempenho de implementações de um algoritmo em diferentes linguagens / compiladores) com benchmarks, gráficos e explicações mais detalhados. Farei imediatamente a reserva de que vi o artigo Oh, esse lento C / C ++ e, provavelmente, se você fizer essas alterações no código C, a imagem mudará um pouco, mas mesmo nesse caso, o python é capaz de exceder a velocidade do C, mesmo nesta versão é em si notável.

Substituiu a lista Python por uma matriz numpy (e, consequentemente, v0[:] por v0.copy() , porque em numpy a[:] retorna a view vez de copiar).
Para entender a natureza do comportamento de desempenho, fiz uma "varredura" pelo número de elementos na matriz.
No código Python, substituí time.monotonic por time.perf_counter , pois é mais preciso (1us versus 1ms para monotônico).
Como o numba usa a compilação jit, essa mesma compilação deve ocorrer algum dia. Por padrão, ocorre na primeira vez em que a função é chamada e afeta inevitavelmente os resultados dos benchmarks (embora se você dedicar um tempo de três lançamentos, talvez não perceba isso), e também é sentida no uso prático. Existem várias maneiras de lidar com esse fenômeno:
1) resultados da compilação de cache no disco:
@njit(cache=True) def lev_dist(s1: AnyStr, s2: AnyStr) -> int:
a compilação ocorrerá na primeira chamada do programa e nas chamadas subseqüentes serão extraídas do disco.
2) indicar a assinatura
A compilação ocorrerá no momento em que o python analisa a definição da função e o primeiro início já será rápido.
A string original é transmitida (mais precisamente, bytes), mas o suporte a strings foi adicionado recentemente, portanto a assinatura é bastante monstruosa (veja abaixo). As assinaturas geralmente são escritas de maneira mais simples:
@njit(nb.int64(nb.uint8[:], nb.uint8[:])) def lev_dist(s1, s2):
mas você precisa converter bytes em uma matriz numpy com antecedência:
s1_py = [int(x) for x in b"a" * 15000] s1 = np.array(s1_py, dtype=np.uint8)
ou
s1 = np.full(15000, ord('a'), dtype=np.uint8)
E você pode deixar os bytes como estão e especificar a assinatura neste formulário:
@njit(nb.int64(nb.bytes(nb.uint8, nb.1d, nb.C), nb.bytes(nb.uint8, nb.1d, nb.C))) def lev_dist(s1: AnyStr, s2: AnyStr) -> int:
A velocidade de execução dos bytes e da matriz numpy do uint8 (neste caso) é a mesma.
3) cache de pré-aquecimento
s1 = b"a" * 15
Em seguida, a compilação ocorrerá na primeira chamada e a segunda já será rápida.
Código C (clang -O3 -march = nativo) #include <stdio.h> #include <stdlib.h> #include <string.h> #include <time.h> static long lev_dist (const char *s1, unsigned long m, const char *s2, unsigned long n) { // unsigned long m, n; unsigned long i, j; long *v0, *v1; long ret, *temp; /* Edge cases. */ if (m == 0) { return n; } else if (n == 0) { return m; } v0 = malloc (sizeof (long) * (n + 1)); v1 = malloc (sizeof (long) * (n + 1)); if (v0 == NULL || v1 == NULL) { fprintf (stderr, "failed to allocate memory\n"); exit (-1); } for (i = 0; i <= n; ++i) { v0[i] = i; } memcpy (v1, v0, sizeof(long) * (n + 1)); for (i = 0; i < m; ++i) { v1[0] = i + 1; for (j = 0; j < n; ++j) { const long subst_cost = (s1[i] == s2[j]) ? v0[j] : (v0[j] + 1); const long del_cost = v0[j + 1] + 1; const long ins_cost = v1[j] + 1; #if !defined(__GNUC__) || defined(__llvm__) if (subst_cost < del_cost) { v1[j + 1] = subst_cost; } else { v1[j + 1] = del_cost; } #else v1[j + 1] = (subst_cost < del_cost) ? subst_cost : del_cost; #endif if (ins_cost < v1[j + 1]) { v1[j + 1] = ins_cost; } } temp = v0; v0 = v1; v1 = temp; } ret = v0[n]; free (v0); free (v1); return ret; } int main () { char s1[25001], s2[25001], s3[25001]; int lengths[] = {1000, 2000, 5000, 10000, 15000, 20000, 25000}; FILE *fout; fopen_s(&fout, "c.txt", "w"); for(int j = 0; j < sizeof(lengths)/sizeof(lengths[0]); j++){ int len = lengths[j]; int i; clock_t start_time, exec_time; for (i = 0; i < len; ++i) { s1[i] = 'a'; s2[i] = 'a'; s3[i] = 'b'; } s1[len] = s2[len] = s3[len] = '\0'; start_time = clock (); printf ("%ld\n", lev_dist (s1, len, s2, len)); printf ("%ld\n", lev_dist (s1, len, s3, len)); exec_time = clock () - start_time; fprintf(fout, "%d %.6f\n", len, ((double) exec_time) / CLOCKS_PER_SEC); fprintf (stderr, "Finished in %.3fs\n", ((double) exec_time) / CLOCKS_PER_SEC); } return 0; }
A comparação foi realizada no windows (windows 10 x64, python 3.7.3, numba 0.45.1, clang 9.0.0, intel m5-6y54 skylake): e no linux (debian 4.9.30, python 3.7.4, numba 0.45.1, 9.0.0).
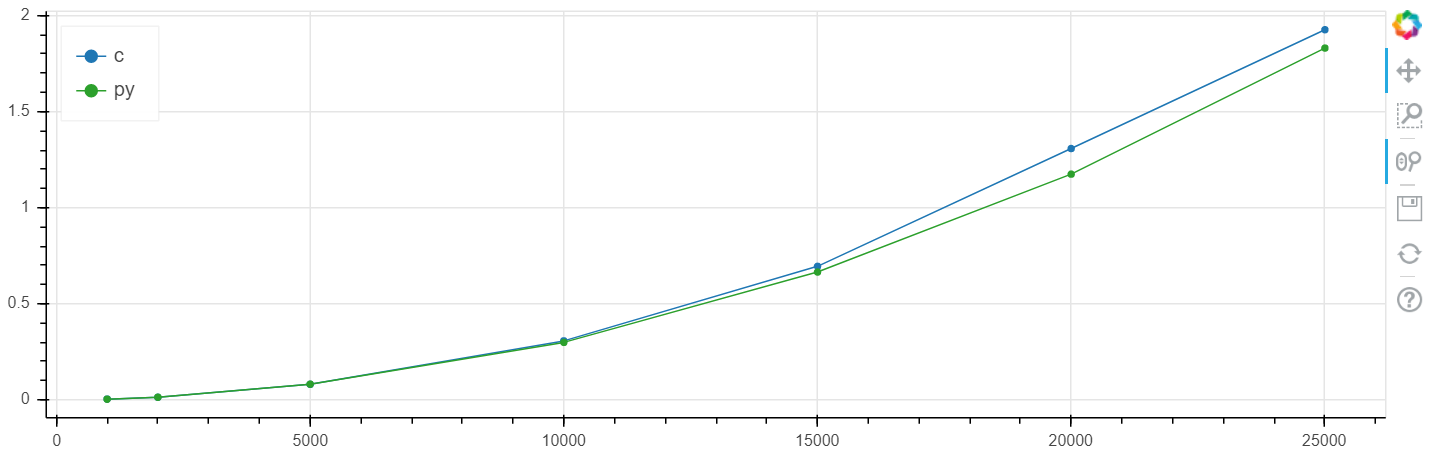
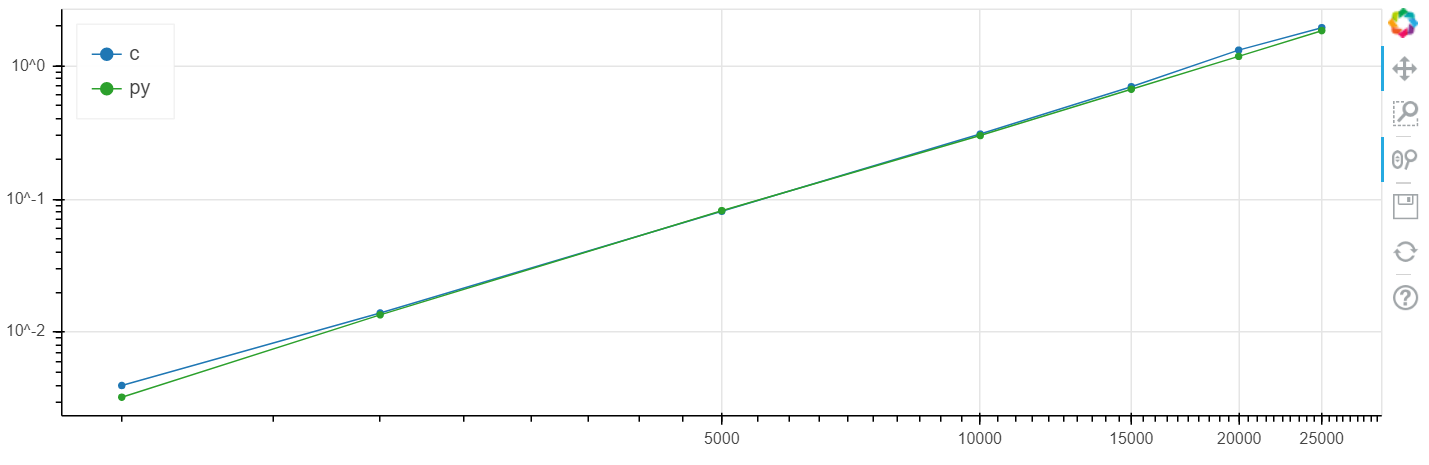
X é o tamanho da matriz, y é o tempo em segundos.
Escala linear do Windows:
Escala logarítmica do Windows:
Escala linear do Linux:

Escala logarítmica do Linux
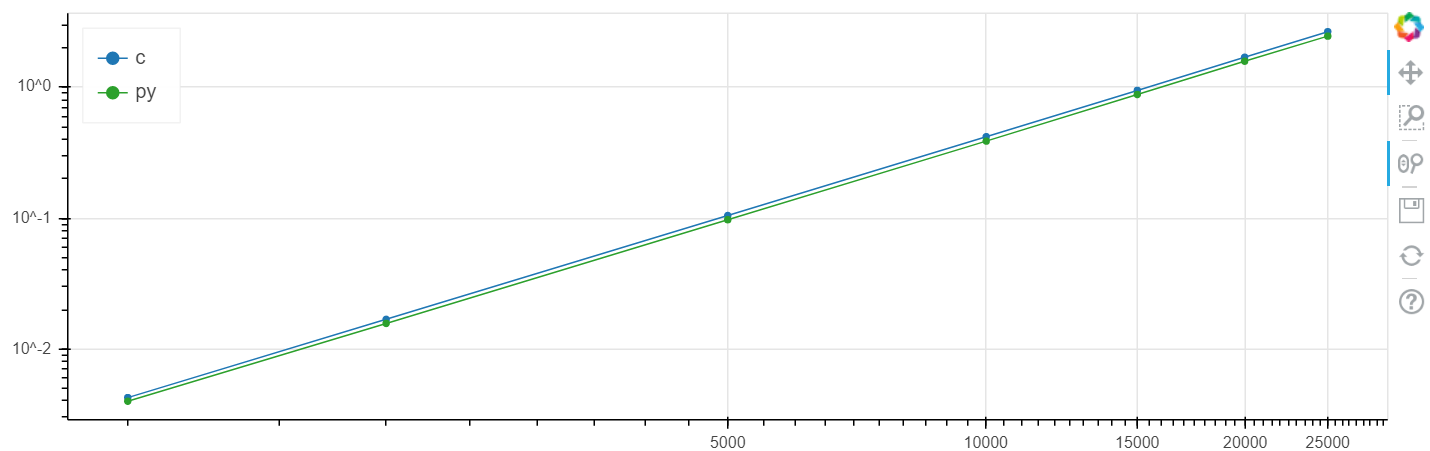
Nesse problema, foi obtido um aumento na velocidade comparado ao clang no nível de vários por cento, o que geralmente é maior que o erro estatístico.
Eu fiz essa comparação repetidamente em diferentes tarefas e, como regra, se o numba pode acelerar algo, ele o acelera a uma velocidade dentro da margem de erro coincidente com a velocidade C (sem o uso de inserções de montagem).
Repito que, se você fizer alterações no código em C de Oh, essa situação lenta do C / C ++ poderá mudar.
Ficarei feliz em ouvir perguntas e sugestões nos comentários.
PS Ao especificar a assinatura de matrizes, é melhor definir explicitamente a maneira de alternar linhas / colunas:
para que a numba não pense em 'C' (si) isso ou 'A' (reconhecimento automático de si / fortran) - por algum motivo, isso afeta o desempenho mesmo para matrizes unidimensionais, por isso existe uma sintaxe tão original: uint8[:,:] this ' A '(detecção automática), nb.uint8[:, ::1] é' C '(si), np.uint8[::1, :] é' F '(fortran).
@njit(nb.int64(nb.uint8[::1], nb.uint8[::1])) def lev_dist(s1, s2):