表观遗传生物标志物的故事始于2013年。 然后是这个方向的先驱者,遗传学和生物统计学领域的专家,加利福尼亚大学洛杉矶分校的史蒂夫·霍瓦斯(Steve Horvath)员工,他介绍了他革命性的确定生物年龄的新方法,称为“表观遗传钟”。 顾名思义,该方法基于表观基因组的变化,即DNA甲基化。
DNA甲基化是调节基因表达的表观遗传机制之一。 在甲基化过程中,CH3特殊酶的甲基与DNA碱基之一胞嘧啶结合。 结果,形成了5-甲基胞嘧啶并使基因表达失活-转录过程被阻断。 众所周知,DNA甲基化是一个动态过程。 它可以在外部因素的影响下发生变化,与许多病理的发展相关,并且可以被多个下一代继承。 甲基化在外源DNA的失活以及发育和衰老过程中起关键作用。 描述了与年龄相关的甲基化变化,称为“表观遗传漂移”。 因此,随着年龄的增长,观察到了低甲基化(去甲基化)和相关的染色体不稳定。 此外,随着年龄的增长,逆过程也会发生-某些启动子区域的甲基化,包括某些肿瘤抑制基因,这与病理学发展有关[1]。 一般而言,今天人们认为甲基化变化在衰老中起关键作用。
基于时间的年龄与整个基因组中许多区域的表观基因组,低甲基和高甲基化的可预测变化相关的事实,已经开发了基于DNA甲基化的第一代表观遗传衰老生物标记。 2011年,加利福尼亚大学洛杉矶分校的科学家发表的第一篇名为“年龄的表观遗传预测因子”的文章表明,DNA甲基化与年龄有着明显的关系。 2013年,史蒂夫·霍瓦特(Steve Horvat)使用计算机辅助的机器学习技术,确定了353个甲基化位点,这些位点与人的年龄高度相关,构成了第一个“表观遗传钟”或“克罗地亚钟”的基础[2]。
这个发现的重要性很难被高估。 必须说,S。Horvat的成功故事充满了未曾发明的戏剧。 根据霍瓦特本人的回忆录,起初,他们不愿意在多家杂志中接受他关于年龄的新表观遗传学方法的文章。 他经常不得不从审稿人那里听到同样的话:“太好了,无法置信。” 但是Horvath并没有放弃,并且全心全意地完成了新方法:“我在每个空闲时间都写了东西,好像这是最后一次完成我的文章。” 在克罗地亚一家人完成工作之前,发生了一场灾难-他刚出生的女儿去世了。 但这并没有使这位固执的科学家感到不安。 他的文章的最新版本再次被基因组生物学拒绝。 在收到审阅者的另一项批评意见之后,霍瓦斯(Horvath)在回忆中做了三件事不应该做的事情:“在阅读了审阅者的评论之后,我花了接下来的10分钟时间做了三件事,这是不应该做的。 首先,我去冰箱,尽可能快地喝了三瓶啤酒。 其次,我回到计算机上并给编辑写了一封信。 第三,我发了。” 而这一次,财富向他微笑,他的文章被接受了。
史蒂夫·霍瓦斯“表观时钟”如何发挥作用? 该发现的作者认为,这种方法在评估抗衰老干预措施的有效性方面可以发挥重要作用,因为它可以大大缩短结果的等待时间。 此外,假设克罗地亚确定的甲基化位点不仅是标记,而且还是衰老的积极参与者(至少一些)。 是什么为研究人员提供了对抗衰老和与年龄相关的病理学的潜在治疗靶标。 同样,当需要从组织样本中提取年龄样本时,这种新方法也可用于刑事诉讼程序。 但是,当然,“表观遗传钟”随后在预测疾病和增加死亡风险方面具有最大的重要性。
事实证明,“表观遗传钟”在胚胎干和诱导的多能细胞,精子,卵细胞和胎盘细胞中显示为“零”。 在生命的最初5年中-与身体发育相关的加速过程。 到21岁时,“表观时钟”逐渐变慢,然后以或多或少的统一速度运行,在外部因素的影响下改变了它的性能。 而且,事实证明,不同的组织以不同的速度衰老。 最容易老化的是女性的乳房组织。 从表观遗传学的角度来看,大脑的衰老速度最慢,而血液和骨骼细胞的衰老则略有加速[3]。
在同一年的2013年,中美两国科学家发表了一篇文章,提出了另一种“表观遗传钟”-“汉纳姆钟”。 加利福尼亚大学圣地亚哥分校的加利福尼亚大学生物工程学教授格雷戈里·汉纳姆(Gregory Hannum)及其同事研究了19至101岁人群血细胞中450,000个CpG二核苷酸的DNA甲基化特征,确定了与年龄最相关的71个甲基化位点[4]。 “ Hannum时钟”和“ Horvat时钟”之间的主要区别在于它们的特异性:DNA样本是从血细胞中获取的,而不是像克罗地亚方法那样从任何组织中获取的。
很快,“表观时钟”的主要价值就变得清晰了。 将他们的进展与按年龄排序比较,对于评估各种原因和多种病理发展导致的死亡风险具有重要的预后价值。 如果“表观时钟”很着急,那就是疾病,加速的衰老和寿命的缩短。 如果他们慢于按时间顺序排列的年龄,就有长寿的好机会。 研究表明,“表观遗传钟”与各种原因和与年龄有关的疾病的死亡率具有高度相关性。2015年,Horvat及其同事进行了研究,以确定肺癌与表观遗传年龄之间的关系。 在分析了2,029人的数据后,发现表观遗传年龄的加快与肺癌风险的增加相关,并且这种关系在吸烟者和70岁以上的人群中更强:“结果表明表观遗传年龄的加速的标准化指标(IEAA)是与肺癌的发生率显着相关(HR:1.50,P = 3.4×10 -3)。 此外,我们已经表明,老年人(70岁或以上)或当前吸烟者之间的关联性甚至更高。 总体而言,我们的结果表明,从生物衰老的角度来看,IEAA可以作为评估肺癌易感性的有用生物标志物” [5]。
同年,另一组研究人员建立了“表观遗传年龄”与60岁以上人群因各种原因死亡的风险的关系。 为了进行分析,我们从四项研究(总计4658人)中获取了数据,参与者的平均年龄分别为79.1、69.5、66.3和72.9岁。 表观遗传年龄通过两种方法确定:“克罗地亚钟”(基于353 CpG甲基化位点)和“汉纳钟”(基于71 CpG甲基化位点)。 两种方法都显示出很强的相关性,尽管基本组仅在6个CpGs位点具有匹配。 研究结果表明,与表观年代相比,“表观遗传年龄”的加速增长了5年,使人们死亡的风险增加了16%。 作者得出结论:“源自DNA甲基化的加速衰老是遗传因素,可预测死亡率,而与健康状况,生活方式因素或已知遗传因素无关。 因此,可以假设DNA甲基化的预测年龄是一个“表观遗传时钟”,它测量在一起的生物年龄,但并不总是与时间年龄平行,并且可以给出预期寿命” [6]。
2016年,德国肿瘤学家进行了一项研究,描述了表观遗传加速老化与癌症和心血管疾病以及所有其他原因的死亡率之间的关系。 对于他们的工作,他们使用了Horvath和Hannum的“表观时钟”。 在参加ESTHER研究的1863名老年人中评估了DNA甲基化年龄,其平均年龄为62.5岁。 结果表明,表观遗传年龄大于按时间顺序排列的年龄与较高的死亡率有关。 通过克罗地亚方法确定的“表观遗传年龄”的加速5年导致死亡率增加了22%,而使用汉纳姆方法则增加了16%[7]。
2016年,由史蒂夫·霍瓦斯(Steve Horvath)领导的大型国际研究人员团队进行了大规模的荟萃分析,对来自三个种族/族裔的13089人的数据进行了调查:白人,西班牙裔和非裔美国人。 表观遗传年龄也通过两种方法确定:“克罗地亚钟”和“汉纳姆钟”。 这项工作表明,将“表观遗传时钟”加速一年(与按年龄排序的年龄相比),将各种原因导致的死亡风险提高到4%。 此外,还观察到相反的效果:“表观遗传时间”的放慢导致较低的死亡风险。 此外,研究人员记录了一个有趣的现象:“我们发现5%的人的生物年龄进程更快,从而导致寿命缩短。 在任何成年年龄,加速的衰老会使这些人的死亡风险增加50%。” [8,9]。
影响“表观时钟”进程的许多外部因素也是已知的。 2017年,Horvath及其同事描述了饮食,饮酒,教育和运动与表观遗传时间的关系。 根据作者本人的说法,他们的工作证实了一个众所周知的事实:以蔬菜,鱼类和瘦肉饮食,适量饮酒,体育锻炼和教育会减慢表观遗传的时间,并有助于延长寿命。 胰岛素和葡萄糖,C反应蛋白和甘油三酸酯水平升高,以及体重过多和高血压加速表观基因组的衰老。 在同一项研究中,科学家没有发现抗脱氢药二甲双胍对表观遗传时间的积极作用[10]。 同年,芬兰研究人员再次证明了肥胖与表观遗传加速衰老的关系[11]
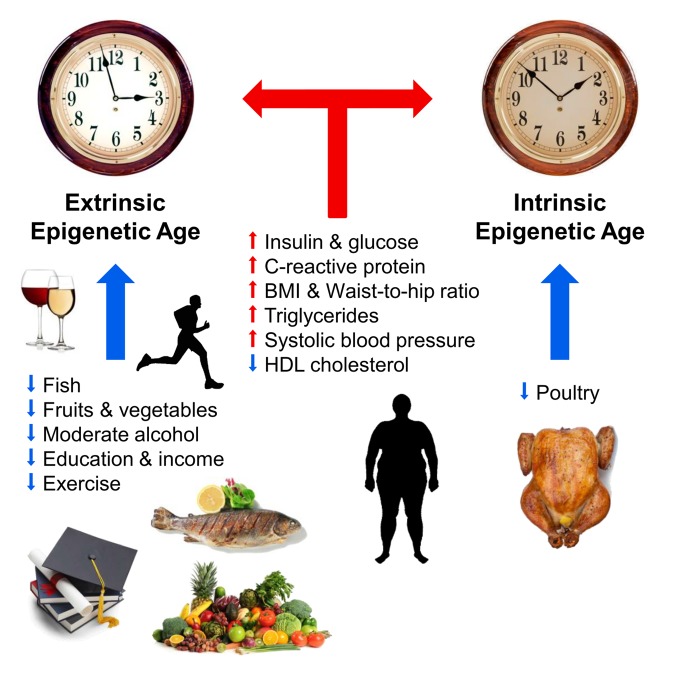
图 1
影响表观遗传年龄的因素 (来自[10])。
蓝色和红色箭头分别表示减缓和加速老化的因素。 时钟象征着外部表观时钟(外部表观时钟,Hannum方法的扩展版本)和内部表观时钟(内部表观时钟,克罗地亚时钟)。 对表观遗传时间过程的积极影响(减慢):鱼类,家禽,蔬菜和水果,高密度脂蛋白,适度酒精,形成和身体活动。 对表观遗传时间的进程产生负面影响(加速):胰岛素,葡萄糖,C反应蛋白,甘油三酸酯,高血压,体重增加以及腰臀比率不正确的水平升高。各种研究小组已经开展了工作,描述了表观遗传时间的过程与身体和认知功能,唐氏和沃纳综合症,HIV感染,阿尔茨海默氏病和更年期之间的关系[12-17]。 以意大利100岁的百岁老人为例,也证明了寿命与“表观遗传钟”减速之间的明确关系[18]。 很长一段时间,史蒂夫·霍瓦斯(Steve Horvath)无法用他的方法来预测哈钦森-吉尔福德(Hutchinson-Guildford)早衰症期间生物学年龄的加速进程。 但是今年这个问题解决了:他和他的同事们基于成纤维细胞创建了特殊的“表观遗传钟”,由391个CpG位点组成,称为“皮血钟” [19]。
S. Horvat工作的下一步是开发更有效的衰老生物标志物,并提高诊断能力,从而预测各种原因导致的死亡风险以及与年龄有关的疾病的发展。 根据霍瓦斯(Horvath)的观点,以前的“表观遗传钟”的缺点是,使用时间年龄作为确定年龄偏差的标准可以排除CpG位点,这些位点的甲基化谱图没有显示出强烈的时间依赖性变化。 相反,它们仅显示了生物学年龄和年代年龄之间的差异。 因此,重要的是,不仅要捕获按时间顺序显示差异的CpG,而且还应捕捉在相同时间顺序的人群中显示风险和生理状态差异的CpG。.2018年,一种称为DNAm PhenoAge的新生物标记已创建。
在第一阶段,S。Horvath团队分析了NHANES III研究的9996人,包括42个临床生物标志物和按年龄划分的年龄与死亡风险之间的关系。 结果,确定了9种生物标志物来预测表型年龄:
1.白蛋白
2.肌酐
3.葡萄糖
4. C反应蛋白
5.淋巴细胞百分比
6.红细胞的平均体积
7.红细胞的分布宽度
8.白细胞计数
9.碱性磷酸酶。然后,他们验证了所选的生物标志物-在另一个样本(6209人)中检查了他们的表型年龄模型。 检查显示表型年龄的增加与各种原因导致的死亡风险增加之间存在相关性:表型年龄一年增加与所有原因导致的死亡风险增加9%,与衰老相关的疾病导致的死亡风险增加9%,心脏病造成的死亡风险增加10%血管疾病,癌症死亡风险增加7%,糖尿病死亡风险增加20%,呼吸系统疾病死亡风险增加9%。
在研究的第二阶段,作者将DNA甲基化与表型年龄相关联。 科学家已经鉴定出哪些甲基化的GpC位点与这9种预测表型年龄的临床生物标志物相关。 他们确定了预测表型年龄的此类位点的513 CpGs。 一种新的表观遗传标记称为DNAm PhenoAge。 该测试显示了新生物标记物的较高准确性:1998年至2007年之间,DNAm PhenoAge的平均变化为8.51年,而临床表型年龄的平均变化为8.88年。
之后,研究人员使用来自4个大型研究的数据测试了他们的新标记生物标志物,这些研究包括妇女健康倡议(n = 4207),弗雷明汉心脏研究(n = 2553),规范性衰老研究(n = 657)和杰克逊心脏研究(n = 1747)。 结果表明,在所有研究中,DNAm PhenoAge与随后的死亡风险均显着相关(不考虑年代年龄):DNAm PhenoAge一年升高与全因死亡风险增加4.5%相关。
DNAm PhenoAge的遗传分析表明,它所使用的513个CpG位点具有与克罗地亚钟相同的41个CpG和与汉纳姆钟相同的6个CpG。 在所有三种表观遗传方法中发现了五个相同的CpG。 来自DNAm PhenoAge的149个CpG位于CpG二核苷酸簇(CpG岛)中。
史蒂夫·霍瓦斯(Steve Horvath)和他的团队在研究过程中获得的所有数据表明,这种新的生物标志物在确定与年龄有关的疾病和死亡的风险方面具有很大的预测价值。 “
通过创新的两步过程,我们开发了一种新的衰老表观遗传生物标记物DNAm PhenoAge,在各种衰老因素(包括各种原因引起的死亡率,癌症,总体健康,身体功能和阿尔茨海默氏病)的预测方面,它已经远远超过了以前的生物标记。 尽管此生物标志物是使用全血数据开发的,但它与每个测试的组织和细胞的年龄密切相关。 根据对分类细胞进行的深入转录分析,我们发现表观遗传年龄相对于时间年龄的增加与促炎和干扰素途径的激活增加以及转录/翻译机制的激活减少,对DNA损伤和线粒体特征的响应降低有关。通常,这种衰老的表观遗传生物标志物能够掩盖在不同组织和细胞中产生多种结果的风险,并提供对衰老重要途径的理解 ” [20]。作者:Alexey Rzheshevsky。参考文献- .., .., .. . . 2011. 42, 1–21;
- Horvath S. DNA methylation age of human tissues and cell types. Genome Biol. 2013. 14, R115.
- Josh Mitteldorf. Methylation Aging Clock: An Update. February 14, 2018.
- Hannum, G; Guinney, J; Zhao, L; Zhang, L; Hughes, G; Sadda, S; Klotzle, B; Bibikova, M; Fan, JB; Gao, Y; Deconde, R; Chen, M; Rajapakse, I; Friend, S; Ideker, T; Zhang, K (2013). Genome-wide methylation profiles reveal quantitative views of human aging rates. Mol Cell. 49: 359–367.
- Morgan E. Levine, H. Dean Hosgood, Brian Chen, Devin Absher, Themistocles Assimes and Steve Horvath. DNA methylation age of blood predicts future onset of lung cancer in the women's health initiative. Aging (Albany NY). 2015 Sep; 7(9): 690–700.
- Riccardo E Marioni, Sonia Shah, et al. DNA methylation age of blood predicts all-cause mortality in later life. Genome Biol. 2015; 16(1): 25.
- Laura Perna, Yan Zhang, Ute Mons, Bernd Holleczek, Kai-Uwe Saum, and Hermann Brenner. Epigenetic age acceleration predicts cancer, cardiovascular, and all-cause mortality in a German case cohort. Clin Epigenetics. 2016; 8: 64.
- Brian H. Chen, Riccardo E. Marioni et al. DNA methylation-based measures of biological age: meta-analysis predicting time to death Aging (Albany NY). 2016 Sep; 8(9): 1844–1859.
- Epigenetic clock predicts life expectancy. ScienceDaily. 28 September 2016.
- Quach A1, Levine ME1 et al. Epigenetic clock analysis of diet, exercise, education, and lifestyle factors. Aging (Albany NY). 2017 Feb 14;9(2):419-446.
- Nevalainen T, Kananen L, Marttila S, Jylhävä J, Mononen N, Kähönen M, Raitakari OT, Hervonen A, Jylhä M, Lehtimäki T, Hurme M. Obesity accelerates epigenetic aging in middle-aged but not in elderly individuals. Clin Epigenetics. 2017 Feb 14;9:20.
- Marioni RE, Shah S, McRae AF, Ritchie SJ, Muniz-Terrera G, Harris SE, Gibson J, Redmond P, Cox SR, Pattie A, Corley J, Taylor A, Murphy L, et al.. The epigenetic clock is correlated with physical and cognitive fitness in the Lothian Birth Cohort 1936. Int J Epidemiol. 2015; 44:1388–96.
- Horvath S, Garagnani P, Bacalini MG, Pirazzini C, Salvioli S, Gentilini D, Di Blasio AM, Giuliani C, Tung S, Vinters HV, Franceschi C. Accelerated epigenetic aging in Down syndrome. Aging Cell. 2015; 14:491–95.
- Horvath S, Levine AJ. HIV-1 Infection Accelerates Age According to the Epigenetic Clock. J Infect Dis. 2015; 212:1563–73.
- Maierhofer A, Flunkert J, Oshima J, Martin GM, Haaf T, Horvath S. Accelerated epigenetic aging in Werner syndrome. Aging (Albany NY). 2017; 9:1143–52.
- Levine ME, Lu AT, Bennett DA, Horvath S. Epigenetic age of the pre-frontal cortex is associated with neuritic plaques, amyloid load, and Alzheimer's disease related cognitive functioning. Aging (Albany NY). 2015; 7:1198–211
- Levine ME, Lu AT, Chen BH, Hernandez DG, Singleton AB, Ferrucci L, Bandinelli S, Salfati E, Manson JE, Quach A, Kusters CD, Kuh D, Wong A, et al.. Menopause accelerates biological aging. Proc Natl Acad Sci USA. 2016; 113:9327–32.
- Horvath S, Pirazzini C, Bacalini MG, Gentilini D, Di Blasio AM, Delledonne M, Mari D, Arosio B, Monti D, Passarino G, De Rango F, D'Aquila P, Giuliani C, et al.. Decreased epigenetic age of PBMCs from Italian semi-supercentenarians and their offspring. Aging (Albany NY). 2015; 7:1159–70.
- Steve Horvath, Junko Oshima et al. Epigenetic clock for skin and blood cells applied to Hutchinson Gilford Progeria Syndrome and ex vivo studies. Aging. Volume 10, Issue 7, pp 1758—75.
- 20. Morgan E. Levine, Ake T. Lu, et al. An epigenetic biomarker of aging for lifespan and healthspan. Aging (Albany NY). 2018 Apr; 10(4): 573–591.